Richard Dawkins jest dobrze znany jako biolog ewolucyjny i agresywny orędownik ateizmu. Wygłaszał on – między innymi w bestsellerze Bóg urojony1 – różne stanowcze twierdzenia na temat nieistnienia projektanta życia. Mając na uwadze jego stanowisko, należałoby zadać mu pewne pytanie. Naukowcy często formułują przewidywania. Ze względu na gromadzone dane empiryczne przewidywania mogą okazać się trafne lub nietrafne, a to z kolei może mieć poważne konsekwencje. Dawkins wielokrotnie opisywał test, który miał według niego potwierdzać teorię ewolucji. Zważywszy jednak na coraz większy korpus danych empirycznych, ten sam test prowadzi nas do wniosku o inteligentnym projekcie. Czy Dawkins zaakceptuje ten wniosek i stanie się zwolennikiem teorii inteligentnego projektu?
Test sformułowany przez Dawkinsa przedstawia się następująco: twierdzi on, że jeśli darwinowska teoria ewolucji jest słuszna, to na podstawie każdego genu w danej grupie organizmów można uzyskać „w przybliżeniu takie samo drzewo życia”2. Jeśli natomiast słuszna jest teoria inteligentnego projektu, to projektant mógłby „wybierać sobie […] co lepsze geny”3 dla każdego organizmu. W takim przypadku – twierdzi – geny nie ukazywałyby takiego samego drzewa podobieństw genetycznych.
Dawkins zaproponował ten test w wydanej w 2009 roku książce Najwspanialsze widowisko świata. Świadectwa ewolucji. Uznał go za „naprawdę najpotężniejsze świadectwo prawdziwości [teorii] ewolucji”4. W tamtym czasie sądził najwyraźniej, że na podstawie każdego genu rzeczywiście można uzyskać w przybliżeniu takie samo drzewo życia. Napisał:
Dowody z porównania DNA (albo białek) mogą zostać wykorzystane – oczywiście zgodnie ze stanowiskiem ewolucyjnym – do rozstrzygania, które gatunki są ze sobą bliżej spokrewnione. Tym jednak, co czyni je naprawdę najpotężniejszym świadectwem prawdziwości [teorii] ewolucji, jest fakt, że można odtworzyć genetyczne drzewo rodowe dla każdego genu z osobna i że taka procedura prowadzi do uzyskania takich samych rezultatów dla różnych genów. Co więcej, tak uzyskane drzewa rodowe z bardzo dużą dokładnością pokrywają się z autentycznym drzewem rodowym gatunków. Za każdym razem otrzymujemy obraz dokładnie taki, jak przewiduje teoria ewolucji, nie zaś coś, czego można by się spodziewać, gdyby postulowany przez kreacjonistów Projektant wybierał sobie z całego królestwa zwierząt („pożyczał”) co lepsze geny, żeby powkładać je do innych organizmów5.
Dawkins mówił o tym teście wielokrotnie
Dawkins formułował to przewidywanie również przy innych okazjach. Zrobił to na przykład w wywiadzie udzielonym w 2009 roku:
Najbardziej przekonującym faktem lub obserwacją, na którą można wskazać, jest wzorzec podobieństw dostrzegalny, kiedy porównuje się geny […] dowolnej pary zwierząt […] i następnie analizuje się podobieństwa między nimi, tworzące doskonałą hierarchię, doskonałe drzewo rodowe. Jedyną alternatywą jest drzewo rodowe, które inteligentny projektant utworzyłby tak, aby zwieść nas w najbardziej podstępny i oszukańczy sposób6.
O tym samym mówił też w wywiadzie w 2010 roku:
Otrzymujemy takie samo drzewo rodowe. Takie samo drzewo rodowe uzyskujemy również wtedy, gdy zbadamy geny, które nie są już funkcjonalne, czyli są szczątkowe i do niczego nie służą. […] Te świadectwa mają przytłaczającą moc. Jedynym możliwym sposobem odrzucenia tezy, że dowodzi to prawdziwości teorii ewolucji, jest uznanie, że inteligentny projektant, Bóg, celowo chciał nas okłamać czy zwieść7.
Ten test jest prosty. Dawkins zestawił ze sobą dwa konkurencyjne przewidywania: jedno na rzecz teorii ewolucji i jedno na rzecz teorii inteligentnego projektu. Twierdzi on, że teoria ewolucji przewiduje „doskonałą” zgodność między różnymi reprezentacjami drzewa życia – to świadectwo jest według niego tak „potężne”, że „dowodzi prawdziwości teorii ewolucji”. Natomiast niezgodności bądź konflikty między różnymi opartymi na danych genetycznych drzewami uznaje on za przewidywanie „alternatywne” w stosunku do przewidywania teorii ewolucji, czyli przewidywanie na rzecz teorii inteligentnego projektu.
W późniejszych latach naukowcy zsekwencjonowali dużą liczbę całych genomów, dzięki czemu wiedzą, że Dawkins się mylił. To nieprawda, że na podstawie każdego genu uzyskuje się „w przybliżeniu takie samo drzewo życia”.
Temat ten był poruszany przeze mnie i innych już wcześniej8, ale niedawno przemyślałem go ponownie, gdy obejrzałem wykład inauguracyjny profesora genomiki ewolucyjnej z Queen Mary University of London. Wykład nosi intrygujący tytuł: Trees of Life: Do They Exist?[Drzewa życia. Czy one istnieją?]9.
Profesor Richard Buggs wspomina o tym, że naukowcy prowadzący badania filogenetyczne zwykle zakładają istnienie darwinowskiego drzewa życia. W literaturze bardzo rzadko da się znaleźć kogoś, kto rzeczywiście podjąłby próbę dowiedzenia istnienia tego drzewa. Jako przykład tych rzadkich przypadków profesor wskazuje twierdzenia Dawkinsa w książce Najwspanialsze widowisko świata, jak również artykuł opublikowany w 1982 roku na łamach „Nature”10, do którego odwołuje się Dawkins. W trakcie wykładu Buggs przytoczył słowa Dawkinsa, że „na podstawie każdego genu uzyskujemy w przybliżeniu takie samo drzewo życia”, i następująco je skomentował: „Wielu z was, którzy tak jak ja codziennie macie do czynienia z danymi pochodzącymi z analiz sekwencji, prawdopodobnie skrzywiło się, słysząc te słowa, ponieważ wiecie, że w istocie nie jest to prawda”.
W gruncie rzeczy, biorąc za podstawę wyniki własnych badań, Buggs wraz ze współpracownikami napisał artykuł opublikowany na łamach czasopisma „Nature Ecology & Evolution”, zatytułowany Convergent Molecular Evolution Among Ash Species Resistant to the Emerald Ash Borer [Konwergentna ewolucja molekularna pośród gatunków jesionów odpornych na opiętka jesionowca]11. W artykule wskazano, że różne geny prowadzą do bardzo odmiennych drzew genowych pośród blisko spokrewnionych gatunków jesionów. W trakcie wykładu Buggs zauważył, że wprawdzie w artykule opisano drzewo „zgodności”, ale nie oznacza to, że te różne geny pozwalają uzyskać takie samo drzewo. Było to natomiast „najlepsze drzewo, które udało się otrzymać na podstawie wszystkich bardzo rozbieżnych drzew genowych”.
Dawkins mógłby zbagatelizować ten przykład, ponieważ dotyczy on wyłącznie gatunków w obrębie rodzaju, a te gatunki wciąż mogą być zdolne do krzyżowania się między sobą nawzajem. Buggs wskazał jednak na to, że różne geny tworzą odmienne drzewa genowe nawet wówczas, gdy badane gatunki bardzo się od siebie różnią. Podał on przykład artykułu, w którym opisano wyniki badań pokrewieństwa między roślinami dwuliściennymi właściwymi (grupą roślin okrytonasiennych mających dwa liście zarodkowe)12. Cecha ta jest charakterystyczna dla całego drzewa życia. Buggs pokazał diagram zaczerpnięty z tego artykułu, na którym zobrazowano „niezgodności” między różnymi opartymi na danych genetycznych drzewami:
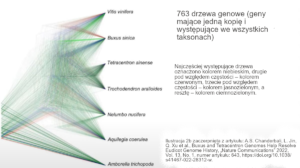
Tak następnie skomentował to profesor Buggs:
W świetle zaproponowanego przez Richarda Dawkinsa testu darwinowskiej koncepcji drzewa życia niewiele więc ją potwierdza, nieprawdaż? Przypomina ona raczej hipotezę, którą Dawkins sformułował na rzecz przekonania o istnieniu inteligentnego projektanta.
Test Dawkinsa w istocie przechylił szalę na korzyść teorii inteligentnego projektu.
Jakie jest więc aktualne stanowisko Richarda Dawkinsa w tej kwestii? Czy wciąż uważa on, że jest to potężny test, który umożliwia rozstrzygnięcie między teorią inteligentnego projektu a darwinowską teorią ewolucji? Czy odrzucił on ten test, czy może jednak przekonał się o prawdziwości teorii inteligentnego projektu? Albo zmienił zdanie na temat sposobu działała ewolucji?
Liczne inne niedawno opublikowane artykuły
Czy mówi o tym wyłącznie profesor Buggs? Nie – również w wielu innych niedawno opublikowanych artykułach wskazuje się na niezgodności między drzewami życia (wszystkie wyróżnienia zostały dodane):
- Dominik Schrempf, Gergely Szöllősi, The Sources of Phylogenetic Conflicts, w: Phylogenetics in the Genomic Era, eds. C. Scornavacca, F. Delsuc, N. Galtier, 2020, chapter No. 3.1, s. 3.1:1–3.1:23 [dostęp 29 VI 2023].
W tym artykule stwierdzono, że „niezgodne sygnały filogenetyczne między genami występują powszechnie” (s. 3.1.1).
- Caroline Parins-Fukuchi, Gregory W. Stull, Stephen A. Smith, Phylogenomic Conflict Coincides with Rapid Morphological Innovation, „Proceedings of the National Academy of Sciences”, 2021, Vol. 118, No. 19, numer artykułu: e2023058118, https://doi.org/10.1073/pnas.2023058118.
W tym artykule zauważono: „Konflikt filogenomowy, polegający na tym, że drzewa genowe są wzajemnie niezgodne w przypadku różnych gatunków, jest powszechny między genomami, jak również w całym Drzewie Życia”.
- Gonzalo Giribet, Genomics and the Animal Tree of Life: Conflicts and Future Prospects, „Zoologica Scripta” 2016, Vol. 45, No. S1, s. 14–21, https://doi.org/10.1111/zsc.12215.
W tym artykule pojawiło się podobne stwierdzenie: „Nowa dziedzina – filogenomika – przynosi nowe kwestie sporne niespotykane wcześniej w ramach filogenetyki molekularnej, mianowicie mające mocne potwierdzenie wzajemnie sprzeczne wyniki” (s. 15).
- Tetyana Nosenko et al., Deep Metazoan Phylogeny: When Different Genes Tell Different Stories, „Molecular Phylogenetics and Evolution” 2013, Vol. 67, No. 1, s. 223–233, https://doi.org/10.1016/j.ympev.2013.01.010.
W tym artykule dokonano przeglądu badań dotyczących pokrewieństw między zwierzętami i znaleziono liczne przykłady niezgodności:
Aby ustalić przyczyny wzorców skrajnych niezgodności u podstawy drzewa życia wielokomórkowców, stworzyliśmy nową supermatrycę zawierającą 122 geny, wzbogaconą o geny nienależące do zwierząt dwubocznie symetrycznych (s. 223). […] Różne matryce genów opowiadają inne historie (s. 226). […] Brak rozwiązania dla głęboko sięgających węzłów na tym drzewie odzwierciedla duże niezgodności między opisanymi już wcześniej filogenezami wielokomórkowców (s. 226). […] najlepiej pasujący model nie pozwolił na ustalenie względnych pozycji kladów zwierząt dwubocznie symetrycznych, jamochłonów oraz płaskowców i gąbek (s. 227). […] Przedstawione tutaj oraz w poprzednich publikacjach liczne wzajemnie niezgodne filogenezy wielokomórkowców łączy jedna cecha wspólna: mają one długie zewnętrzne i krótkie wewnętrzne gałęzie (s. 229). […] Podsumowując, wyniki naszych badań wskazują na trzy wzajemnie niezgodne, ale mające mocne potwierdzenie topologie drzew (s. 230).
- Sushma Reddy et al., Why Do Phylogenomic Data Sets Yield Conflicting Trees? Data Type Influences the Avian Tree of Life more than Taxon Sampling, „Systematic Biology” 2017, Vol. 66, No. 5, s. 857–879, https://doi.org/10.1093/sysbio/syx041.
W tym artykule także zauważono, że naukowcy żywili nadzieję, iż dzięki pobieraniu prób z dużej ilości danych uda im się wyeliminować niezgodności między drzewami. Nadzieja ta okazała się jednak płonna, zwłaszcza w przypadku klasy ptaków:
Filogenomikę, czyli wykorzystywanie wielkoskalowych matryc danych w analizach filogenetycznych, postrzegano jako ostateczne rozwiązanie problemu zrozumienia trudnych węzłów na drzewie życia. Stało się jednak jasne, że analizy tych dużych zbiorów danych genomowych mogą prowadzić do niezgodności w oszacowaniach filogenezy (s. 857). […] Wydawało się, że filogenomika jest wręcz stworzona do spełnienia tej obietnicy rozwiązania problemu drzewa życia. Analizy dużych matryc danych niekiedy prowadziły jednak do wzajemnie niezgodnych topologii, co oznacza, że sam duży zbiór danych nie wystarczy do zrealizowania tego celu (s. 857).
- Cédric Blais, John M. Archibald, The Past, Present and Future of the Tree of Life, „Current Biology” 2021, Vol. 31, No. 7, s. R314–R321, https://doi.org/10.1016/j.cub.2021.02.052.
W tym przeglądzie debaty między zwolennikami i krytykami hipotezy „drzewa życia” przyznano, że drzewa oparte na danych genetycznych często mogą być ze sobą niezgodne, aczkolwiek postuluje się tam również, że pewien wertykalny sygnał pochodzenia nadal jest obecny, mimo iż jest zakłócany przez takie procesy jak horyzontalny transfer genów:
Około dwudziestu lat temu drzewo życia zaczęło się chwiać, gdy uświadomiono sobie, że genomy prokariontów składają się z genów mających odmienne historie ewolucyjne (s. R314). […] Pod koniec lat dziewięćdziesiątych XX wieku eksplozja technik sekwencjonowania całych genomów oraz powstanie genomiki porównawczej miały – jak oczekiwano – potwierdzić i umocnić zaproponowaną przez [Carla] Woesego koncepcję drzewa. Te nadzieje szybko okazały się płonne. Filogenetyka w krótkim czasie uzmysłowiła nam, że różne geny w tym samym genomie mogą prowadzić do bardzo odmiennych topologii drzew i nawet blisko spokrewnione organizmy mają znacznie różniące się zawartości genów. Przywołano nową siłę ewolucyjną: horyzontalny transfer genów, czyli wymianę materiału genetycznego między różnymi gatunkami (s. R315). […]
Opracowanie statystycznego drzewa życia unifikującego wszystkie istniejące gatunki wciąż może być realne, co przeczy oczekiwaniu, że tylko lokalne wzorce drzew przetrwałyby wszechobecny horyzontalny transfer genów. Takie drzewo nie stanowi już jednak kompletnej, całościowej reprezentacji genetycznej historii organizmów, ponieważ nie odpowiada historii żadnego pojedynczego genu. Jeśli koncepcja drzewa życia wciąż jest do utrzymania, to wyłącznie częściowo (s. R318). […] To, że nadal posługujemy się koncepcją drzewa życia w celach klasyfikacji, świadczy więc nie tylko o jej dogodności w praktyce oraz o historycznej i kulturowej inercji, ale też o przywiązaniu do klasyfikacji naturalnej (s. R318). […]
Wydobycie sygnału filogenetycznego z danych genomowych może być trudne, a duża część świadectw na rzecz pokrewieństw między dawnymi organizmami jest w najlepszym razie niekonkluzywna. Metody filogenetyczne opracowane w celu skonstruowania drzewa będą skuteczne niezależnie od tego, czy najlepiej pasują do danych empirycznych. Natomiast koncepcja sieci ujmuje niezgodności między danymi genomowymi bez narzucania interpretacji (s. R319).
- Juli Berwald, The Web of Life: Classic Evolutionary Theory Holds That Species Separate over Time. But It’s Fuzzier Than That – Now We Know They Also Merge, „Aeon” 2022, April 5 [dostęp 1 VII 2023].
W tym artykule przytoczono wypowiedź genetyka Rasmusa Nielsena z Uniwersytetu Kalifornijskiego w Berkeley: „Zawsze wiedzieliśmy, że ten abstrakcyjny pomysł, iż ewolucja tworzy wzorzec drzewa, jest w pewnej mierze nieadekwatny. […] Teraz wiemy jednak, że jest on naprawdę nieadekwatny”.
Nielsen dodał: „Sądzę, że proces rozdzielania się i ponownego łączenia ze sobą, a także wymiany fragmentów DNA między różnymi organizmami zachodzi nieustannie w całym drzewie życia[…]. Obecnie mocno zmienia się nasz sposób patrzenia na tę sprawę, uważamy bowiem, że mamy do czynienia z siecią, nie zaś z drzewem życia”.
W artykule uznano więc, że historia życia tworzy wzorzec sieciowy, a nie wzorzec drzewa: „Zgodnie z hipotezą sieciowej ewolucji gatunki nie są odizolowane od siebie nawzajem, jak wskazuje haecklowska koncepcja rozgałęziającego się drzewa. Gatunki zarówno ulegają dywersyfikacji, jak i łączą się ze sobą nawzajem. Drzewo życia nie wygląda więc jak drzewo, lecz bardziej przypomina wzór siatkowy widoczny na skórze pytona”.
Zjawisko pojawiania się genów w miejscach nieoczekiwanych przez tradycyjną koncepcję wertykalnej wspólnoty pochodzenia występuje bardzo często, ponieważ „Wędrujące geny odkryto w każdej gałęzi drzewa życia, którą badali genetycy”. W artykule postawiono następujące pytania: „Jeśli gatunki trudno usytuować na końcach gałęzi drzew, to co to oznacza dla haecklowskiego modelu drzewa ewolucyjnego? Czy powinniśmy ten model odrzucić?”.
W artykule przyjrzano się wynikom badań pokrewieństw między rybami i uznano, że „Genetycy badający tę dużą grupę długo głowili się nad jej historią ewolucyjną, a w ramach różnych badań ustalano odmienne, ewidentnie wzajemnie niezgodne pokrewieństwa”.
Artykuł kończy się stwierdzeniem, że idea doskonałych drzew ewolucyjnych nie jest słuszna: „Co by się stało, gdybym ponownie miała szansę przemawiać do swoich studentów? Pokazałabym im haecklowskie drzewo na ekranie i powiedziała, że to anachronizm”.
Przetestowanie testu
Zanim zakończę, muszę powiedzieć coś o zasadności zaproponowanego przez Dawkinsa testu teorii inteligentnego projektu. Pod pewnymi względami test ten jest wątpliwy, ale pod innymi ma pewien sens. Dawkins ma prawdopodobnie rację, że teoria inteligentnego projektu nie jest najlepszym wyjaśnieniem „doskonałej” zgodności między drzewami ewolucyjnymi. Tyle że teoria inteligentnego projektu nie twierdzi, że wszystko musi być skutkiem projektu. Doskonała zgodność filogenetyczna wskazuje co najwyżej na wspólnotę pochodzenia, a idea wspólnoty pochodzenia jest akceptowana przez niektórych teoretyków projektu, ponieważ pierwotny projekt mógł być zawarty we wspólnym przodku!
Dawkins niesłusznie mógłby również twierdzić, że dowolny stopień drzewopodobnego rozkładu genów obalałby teorię inteligentnego projektu. To dlatego że zaprojektowane struktury pełnią różne funkcje, a funkcjonalne składniki często są zależne od innych funkcjonalnych składników i oddziałują z nimi w logiczne, funkcjonalnie konieczne sposoby. Może to prowadzić do nielosowych korelacji i hierarchicznych rozkładów cech – czyli efektem projektu mogą być drzewopodobne struktury w zbiorze danych!
Kiedy na przykład widzimy guziki na koszuli, to znajdziemy również otwory na guziki. Kiedy widzimy koła w samochodzie, to niemal zawsze znajdziemy też osie. Takie korelacje mogą tworzyć drzewopodobny rozkład cech. Fizyk Brian Miller wyjaśnia tę koncepcję w niedawno wydanym tomie Science and Faith in Dialogue [Dialog nauki i wiary]:
Architekturom projektowym często odpowiada wzorzec hierarchiczny. Wszystkie pojazdy transportowe posiadają pewne cechy wspólne, takie jak wydzielone miejsca dla towaru lub ludzi, układ napędowy i układ kierowniczy. Oprócz tych cech samochody mają takie elementy, jak koła, hamulce, płyny chłodzące, substancje smarne i osie. Modele Toyoty Camry mają też dodatkowe wyspecjalizowane elementy. Podobieństwa między pojazdami transportowymi najprawdopodobniej pasowałyby do skonstruowanego wzorca drzewa co najmniej równie dobrze jak różne grupy gatunków13.
Między cechami biologicznymi niewątpliwie istnieje wiele analogicznych korelacji – dzięki temu otrzymamy pewną drzewopodobną strukturę w zbiorze danych. Na przykład genom dla nosów u ssaków będą prawdopodobnie towarzyszyć geny dla włosów w nosie, a geny dla palców u lądowych czworonogów często są skorelowane z genami dla pazurów! Istoty inteligentne z reguły nie muszą jednak tworzyć rozkładów cech układających się w zagnieżdżoną hierarchię. Jak wyjaśnia Brian Miller, sensownym oczekiwaniem hipotezy projektu jest zarówno występowanie pewnych drzewopodobnych struktur, jak i licznych niedrzewopodobnych rozkładów cech:
Chociaż wiele cech wytworów człowieka pasuje do hierarchicznego, drzewopodobnego wzorca, to liczne inne nie wpisują się w ten wzorzec. Radiowozy i samoloty są wyposażone w urządzenia nadawczo-odbiorcze, ale w większości innych pojazdów ich nie ma. Ponadto w bardzo różnorodnych pojazdach występuje taki sam zespół obwodów elektrycznych mający realizować podobne cele. Ten wzorzec jest odzwierciedleniem faktu, że inżynierowie często tworzą moduły, które można wykorzystać w różnych kontekstach. Te moduły muszą być projektowane z wyraźnym celem funkcjonowania w różnych produktach, a produkty wykorzystujące te moduły muszą być projektowane tak, aby moduły mogły należycie w nich funkcjonować. Taki sam wzorzec oraz wymogi obserwujemy w przypadku życia14.
Informatyk Winston Ewert opracował nawet model umożliwiający zrozumienie „wspólnego projektu” za pomocą grafów zależności15.
Zauważmy ponadto, że w wywiadzie udzielonym w 2010 roku Richard Dawkins mówił o „genach, które nie są już funkcjonalne”, czyli o tak zwanych pseudogenach. Argumentował on, że istnienie pseudogenów obala teorię inteligentnego projektu, ponieważ żaden projektant nie umieściłby identycznych niefunkcjonalnych genów w tych samych miejscach genomów różnych gatunków. Argument ten jest jednak słaby, zważywszy na naszą wiedzę, że wiele pseudogenów pełni pewne funkcje16. Współdzielone pseudogeny, które naprawdę są niefunkcjonalne, najprawdopodobniej lepiej jest wyjaśniać jako skutek wspólnoty pochodzenia, nie zaś wspólnego projektu. Nawet jeśli jednak obecnie są one niefunkcjonalne, to możliwe, że takie pseudogeny pierwotnie były funkcjonalne, ale ich funkcje uległy z upływem czasu degradacji i te pseudogeny „nie są już funkcjonalne”.
Tutaj projekt i wspólnota pochodzenia również mogą się przecinać: współdzielone pseudogeny – o ile naprawdę są niefunkcjonalne – mogły być pierwotnie zaprojektowane jako elementy funkcjonalne, ale najprawdopodobniej zostały zaprojektowane we wspólnym przodku, nie zaś w oddzielnych gatunkach. Spostrzeżenie Dawkinsa na temat pseudogenów nie stanowi ogólnego testu hipotezy projektu, lecz jedynie test umożliwiający rozstrzygnięcie między koncepcją oddzielnego, choć wspólnego, projektu a koncepcją wspólnoty pochodzenia (która i tak może potencjalnie być rezultatem projektu).
Co teraz powie Richard Dawkins?
W każdym razie to prawda, że przytaczane przeze mnie artykuły nie zostały napisane z zamiarem potwierdzenia teorii inteligentnego projektu. Potwierdzają one jednak, że często występują niezgodności między drzewami filogenetycznymi, jak również że istnieją dane nietworzące wzorców drzewopodobnych między różnymi genami i różnymi gatunkami. Ten wniosek nie wypływa wyłącznie z dawno już opublikowanych artykułów lub artykułów opisujących wyniki badań dotyczących wierzchołków drzewa życia, lecz z artykułów opublikowanych niedawno i opisujących wyniki badań kluczowych aspektów drzewa życia takich jak fundamentalne pokrewieństwa między zwierzętami, roślinami lub mikroorganizmami. A często występujące niezgodności między drzewami filogenetycznymi to właśnie coś, co według Dawkinsa przewiduje teoria inteligentnego projektu.
Dawkins sformułował test mający umożliwiać rozstrzygnięcie między darwinowską teorią ewolucji a teorią inteligentnego projektu. Musi mieć on stale w pamięci swoje wielokrotnie powtarzane twierdzenia. Czy Dawkins przyzna, że jego przewidywanie okazało się nietrafne w przypadku teorii ewolucji, a trafne w odniesieniu do teorii inteligentnego projektu?
Casey Luskin
Oryginał: Intelligent Design Passes the Dawkins Test , „Evolution News & Science Today” 2023, May 15 [dostęp 9 VIII 2023].
Przekład z języka angielskiego: Dariusz Sagan
Źródło zdjęcia: Pixabay
Ostatnia aktualizacja strony: 9.8.2023
Przypisy
- Por. R. Dawkins, Bóg urojony, tłum. P.J. Szwajcer, Wydawnictwo CiS, Warszawa 2007 (przyp. tłum.).
- R. Dawkins, Najwspanialsze widowisko świata. Świadectwa ewolucji, tłum. P.J. Szwajcer, Wydawnictwo CiS, Stare Groszki 2010, s. 393. Zastosowałem tutaj własne tłumaczenie, ponieważ treść istniejącego przekładu na język polski („taka procedura prowadzi do uzyskania takich samych rezultatów dla różnych genów”) nie pasuje w tym miejscu do kontekstu artykułu Caseya Luskina (przyp. tłum.).
- Tamże, s. 394 (przyp. tłum.).
- Tamże, s. 393 (przyp. tłum.).
- Tamże, s. 393–394 [wyróżnienia dodane].
- Richard Dawkins: One Fact to Refute Creationism [dostęp 27 VI 2023] [wyróżnienia dodane] (przyp. tłum.).
- Richard Dawkins Answers Reddit Questions [dostęp 27 VI 2023] [wyróżnienia dodane] (przyp. tłum.).
- Por. np. C. Luskin, Dawkins: A “Perfect Hierarchy” in the Tree of Life?, „Evolution News & Science Today” 2021, September 3 [dostęp27 VI 2023] (przyp. tłum.).
- Por. Trees of Life: Do They Exist? Prof. Richard Buggs Inaugural Lecture [dostęp 27 VI 2023] (przyp. tłum.).
- Por. D. Penny, L.R. Foulds, M.D. Hendy, Testing the Theory of Evolution by Comparing Phylogenetic Trees Constructed from Five Different Protein Sequences, „Nature” 1982, Vol. 297, No. 5863, s. 197–200, https://doi.org/10.1038/297197a0 (przyp. tłum.).
- Por. L.J. Kelly et al., Convergent Molecular Evolution Among Ash Species Resistant to the Emerald Ash Borer, „Nature Ecology & Evolution” 2020, Vol. 4, s. 1116–1128, https://doi.org/10.1038/s41559-020-1209-3.
- Por. A.S. Chanderbali al., Buxus and Tetracentron Genomes Help Resolve Eudicot Genome History, „Nature Communications” 2022, Vol. 13, No. 1, numer artykułu: 643, https://doi.org/10.1038/s41467-022-28312-w.
- B. Miller, Engineering Principles Better Explain Biological Systems Than Evolutionary Theory, w: Science and Faith in Dialogue, eds. F. van Niekerk, N. Vorster, „Reformed Theology in Africa Series”, Vol. 10, AOSIS Books, Cape Town 2022, s. 202–203 [175–211] [dostęp 2 VII 2023] (przyp. tłum.).
- Tamże, s. 203 (przyp. tłum.).
- Por. W. Ewert, The Dependency Graph of Life, „BIO-Complexity” 2018, No. 3, s. 1–27, https://doi.org/10.5048/BIO-C.2018.3.c (przyp. tłum.).
- Por. C. Luskin, Pseudogenes Aren’t Nonfunctional Relics that Refute Intelligent Design, „Evolution News & Science Today” 2021, September 9 [dostęp 2 VII 2023] (przyp. tłum.).
Literatura:
- Berwald J., The Web of Life: Classic Evolutionary Theory Holds That Species Separate over Time. But It’s Fuzzier Than That – Now We Know They Also Merge, „Aeon” 2022, April 5 [dostęp1 VII 2023].
- Blais C., Archibald J.M., The Past, Present and Future of the Tree of Life, „Current Biology” 2021, Vol. 31, No. 7, s. R314–R321, https://doi.org/10.1016/j.cub.2021.02.052.
- Chanderbali A.S. et al., Buxus and Tetracentron Genomes Help Resolve Eudicot Genome History, „Nature Communications” 2022, Vol. 13, No. 1, numer artykułu: 643, https://doi.org/10.1038/s41467-022-28312-w.
- Dawkins R., Bóg urojony, tłum. P.J. Szwajcer, Wydawnictwo CiS, Warszawa 2007.
- Dawkins R., Najwspanialsze widowisko świata. Świadectwa ewolucji, tłum. P.J. Szwajcer, Wydawnictwo CiS, Stare Groszki 2010.
- Ewert W., The Dependency Graph of Life, „BIO-Complexity” 2018, No. 3, s. 1–27, https://doi.org/10.5048/BIO-C.2018.3.c.
- Giribet G., Genomics and the Animal Tree of Life: Conflicts and Future Prospects, „Zoologica Scripta” 2016, Vol. 45, No. S1, s. 14–21, https://doi.org/10.1111/zsc.12215.
- Kelly L.J. et al., Convergent Molecular Evolution Among Ash Species Resistant to the Emerald Ash Borer, „Nature Ecology & Evolution” 2020, Vol. 4, s. 1116–1128, https://doi.org/10.1038/s41559-020-1209-3.
- Luskin C., Dawkins: A “Perfect Hierarchy” in the Tree of Life?, „Evolution News & Science Today” 2021, September 3 [dostęp 27 VI 2023].
- Luskin C., Pseudogenes Aren’t Nonfunctional Relics that Refute Intelligent Design, „Evolution News & Science Today” 2021, September 9 [dostęp 2 VII 2023].
- Miller B., Engineering Principles Better Explain Biological Systems Than Evolutionary Theory, w: Science and Faith in Dialogue, eds. F. van Niekerk, N. Vorster, „Reformed Theology in Africa Series”, Vol. 10, AOSIS Books, Cape Town 2022, s. 175–211 [dostęp 2 VII 2023].
- Nosenko T. et al., Deep Metazoan Phylogeny: When Different Genes Tell Different Stories, „Molecular Phylogenetics and Evolution” 2013, Vol. 67, No. 1, s. 223–233, https://doi.org/10.1016/j.ympev.2013.01.010.
- Parins-Fukuchi C., Stull G.W., Smith S.A., Phylogenomic Conflict Coincides with Rapid Morphological Innovation, „Proceedings of the National Academy of Sciences”, 2021, Vol. 118, No. 19, numer artykułu: e2023058118, https://doi.org/10.1073/pnas.2023058118.
- Penny D., Foulds L.R., Hendy M.D., Testing the Theory of Evolution by Comparing Phylogenetic Trees Constructed from Five Different Protein Sequences, „Nature” 1982, Vol. 297, No. 5863, s. 197–200, https://doi.org/10.1038/297197a0.
- Reddy S. et al., Why Do Phylogenomic Data Sets Yield Conflicting Trees? Data Type Influences the Avian Tree of Life more than Taxon Sampling, „Systematic Biology” 2017, Vol. 66, No. 5, s. 857–879, https://doi.org/10.1093/sysbio/syx041.
- Richard Dawkins Answers Reddit Questions [dostęp 27 VI 2023].
- Richard Dawkins: One Fact to Refute Creationism [dostęp 27 VI 2023].
- Schrempf D., Szöllősi G., The Sources of Phylogenetic Conflicts, w: Phylogenetics in the Genomic Era, eds. C. Scornavacca, F. Delsuc, N. Galtier, 2020, chapter No. 3.1, s. 3.1:1–3.1:23, https://hal.science/hal-02535482 [dostęp 29 VI 2023].
- Trees of Life: Do They Exist? Prof. Richard Buggs Inaugural Lecture [dostęp 27 VI 2023].

