Niniejszy tekst stanowi czwartą część1 w serii artykułów Davida Ussery’ego zatytułowanej Czy istnieje granica ewolucji? Autor tę serię artykułów adresuje zarówno do tych, którzy jeszcze nie przeczytali książki Behego, jak i do tych, którzy już ją znają. Ussery szczegółowo analizuje argumenty przedstawione w książce Michaela J. Behego Granica ewolucji. Uważnie przygląda się pierwszym dziewięciu rozdziałom publikacji Behego oraz wskazuje mocne i słabe strony zawartych w nich argumentów. Krótko przedstawia również swoje osobiste i filozoficzne poglądy.
Rozdział 6 – Punkty odniesienia
Ten rozdział książki Michaela J. Behego Granica ewolucji. W poszukiwaniu ograniczeń darwinizmu2 dotyczy sposobu, w jaki Behe rozstrzyga, czy wytworzenie jakichś cech biologicznych w wyniku losowych mutacji i doboru naturalnego jest mało prawdopodobne. Jako przykład wybrał on cytat z artykułu, w którym mowa o ocenianiu proponowanych mechanizmów biologicznej specjacji na podstawie tego, co wydaje się „biologicznie sensowne”. Behe twierdzi, że idei „biologicznej sensowności” nie sprawdzono w odniesieniu do całej ewolucji, i zamierza dokonać tego właśnie w rozdziale Punkty odniesienia. Aby „rozstrzygnąć, czy losowe mutacje w połączeniu z doborem naturalnym mogą stanowić biologicznie sensowne wyjaśnienie danego zjawiska molekularnego”3, posługuje się on dwoma kryteriami: liczbą kroków koniecznych do wytworzenia czegoś oraz spójnością – uporządkowaniem kroków ze względu na cel. Richard Dawkins spełnia te kryteria w książce Wspinaczka na szczyt nieprawdopodobieństwa4. Byłem zaskoczony, gdy zauważyłem, że mimo iż Behe wspomina Karola Darwina, Daniela C. Dennetta, Johna Maynarda Smitha, H. Allena Orra, Jerry’ego A. Coyne’a i François Jacoba, nie pisze ani słowem o tej klasycznej książce Dawkinsa, w której autor rozprawia się właśnie z takimi argumentami, jak przedstawione w tym rozdziale. Warto dodać, że Wspinaczka na szczyt nieprawdopodobieństwa Dawkinsa ukazała się w 1996 roku, mniej więcej w tym samym czasie co Czarna skrzynka Darwina5 Behego. Myślę, że Dawkins ma słuszność, gdy w swojej recenzji Granicy ewolucji uznaje, że w odróżnieniu od pierwszej książki Behego (Czarna skrzynka Darwina):
Drugą książkę napisał człowiek, który się poddał. Złapany w pułapkę niewłaściwej ścieżki, którą obrał w swoim raczej nieinteligentnym projekcie, Behe nie pozostawił sobie żadnej drogi ucieczki. Ten wzór do naśladowania dla kreacjonistów na całej kuli ziemskiej zupełnie odciął się od świata autentycznej nauki6.
W tym rozdziale Behe dochodzi do wniosku, że ewolucja jest majsterkowiczem, a nie inżynierem. W porządku. Biochemik ten wnioskuje również jednak, że „gdyby ewolucja darwinowska była majsterkowiczem, to nie można by oczekiwać, że wytworzy spójne struktury z wieloma odrębnymi częściami współdziałającymi ze sobą w jasno określonym celu, którego realizacja wymaga udziału większej liczby elementów”7. Co jednak ze Wspinaczką na szczyt nieprawdopodobieństwa Dawkinsa? Co z klasycznym przykładem oka?8 Tego tematu dotyczy wiele książek i artykułów naukowych. Zachęcam zainteresowanego czytelnika do odwiedzenia na przykład strony Amazom.com i przyjrzenia się kilku opublikowanym książkom, które są poświęcone zagadnieniu ewolucji oczu zwierząt9. Można tam znaleźć dokładnie to, co według Behego jest niemożliwe, czyli jasne, szczegółowe opisy powolnej, stopniowej ewolucji takich złożonych układów jak oko. Według mnie (biologa molekularnego) ewolucja oka niewiele różni się od ewolucji złożonego układu biochemicznego. Niewątpliwie inna jest skala obu zjawisk, ale zachodzą one na tych samych zasadach. Proszę jednak nie wierzyć mi na słowo, tylko wpisać w bazie danych PubMed słowa „evolution complex systems” i samemu przekonać się, co da się tam znaleźć.
Rozdział 7 – Reguła dwóch miejsc wiązania
W tym rozdziale Behe kontynuuje swój argument z niedowierzania. Tym razem jednak nie analizuje pojedynczych mutacji w pojedynczych genach, lecz koncentruje się na prawdopodobieństwie wytworzenia przez mechanizmy ewolucyjne dwóch różnych białek mających pasujące do siebie kształty, czyli posiadających komplementarne „miejsca wiązania”. Zastanawia się, jakie są szanse na wyewoluowanie DWÓCH miejsc wiązania w tym samym czasie. Według niego w tym przypadku prawdopodobieństwo jest na tyle nikłe, że taka ewolucja jest w praktyce niemożliwa. Ponownie można jednak mieć zastrzeżenia do wyjściowych założeń przyjętych przez Behego. Nie lubię brzmieć jak zdarta płyta, ale po raz kolejny zachęcam zainteresowanego czytelnika do dokonania przeglądu ogromnej literatury w tej dziedzinie. W bazie danych PubMed wpisałem słowa „evolution protein binding sites” i znalazłem ponad 5000 artykułów. Tytuł jednego z najnowszych [z perspektywy 2010 roku] artykułów brzmi Using Peptide Loop Insertion Mutagenesis for the Evolution of Proteins10 [Wykorzystanie mutagenezy insercji pętli peptydowej do ewolucyjnego tworzenia białek], a innego – Beauty Is in the Eye of the Beholder: Proteins Can Recognize Binding Sites of Homologous Proteins in More Than One Way11 [Piękno jest kwestią gustu. Białka potrafią rozpoznawać miejsca wiązania homologicznych białek na więcej niż jeden sposób]12. Dostrzegam tutaj pewną słabą stronę (z wielu) jego argumentacji w rozdziale 7: istnieje duża przestrzeń dla zmian miejsc wiązania – stuprocentowe dopasowanie nie jest konieczne. Musi być jedynie właściwy kształt, a można go uzyskać za pomocą bardzo wielu różnych sekwencji aminokwasów. Prawdopodobieństwo nie jest więc aż tak znikomo małe, jak można by się spodziewać na podstawie naiwnych i błędnych pierwszych przybliżeń.
Pod koniec tego rozdziału Behe omawia badania Richarda E. Lenskiego z Uniwersytetu Stanu Michigan. Behe twierdzi, że pomimo hodowania bakterii Escherichia coli przez ponad 40 tysięcy pokoleń, „zasadniczo nie powstało nic nowego”13. Znam Richa Lenskiego od wielu lat, a ostatnio przyjechał nawet na mój uniwersytet, by wziąć udział w obronie doktorskiej. Rich miał wspaniałe przemówienie, w trakcie którego pokazał, że na wczesnych etapach swoich eksperymentów zaobserwował wyraźny, mierzalny wzrost stopnia przystosowania związany z nastąpieniem (losowych) mutacji w ramach jego eksperymentów ewolucyjnych. Na przykład w wielu szczepach nastąpił zespół mutacji zmieniających topologię (trójwymiarową strukturę) DNA, a skutkiem tego był wzrost stopnia przystosowania. (Zagadnienie topologii DNA leży w kompetencjach zarówno Behego, jak i moich – szkoda, że Behe nie pracuje już w laboratorium i nie bada struktur oraz ewolucji DNA!). W niektórych późniejszych eksperymentach Lenskiego, po namnażaniu komórek przez ponad piętnaście lat (!), powstał szczep wykazujący zwiększone tempo mutacji. W następstwie tego częstość nowo generowanych mutacji oraz różnorodność radykalnie wzrosła. Wcześniej, w przypadku pierwszych 20 tysięcy pokoleń (co wymagało dziesięciu lat namnażania komórek w laboratorium), liczba utrwalonych zmian genetycznych była – średnio rzecz biorąc – niewielka (zwykle mniej niż dziesięć). Jednak po powstaniu tego szczepu „mutatora” liczba utrwalonych mutacji (nowych wariantów genetycznych, które były obecne we wszystkich komórkach) wzrosła do ponad 250, a liczba pojedynczych zmian wzrosła w sumie do ponad tysiąca14.
W moim przekonaniu wyniki uzyskane przez Lenskiego świetnie podsumowują to, co obserwujemy w sekwencjach genomów. Szczepy laboratoryjne nie cechują się zbyt dużą różnorodnością w porównaniu ze szczepami występującymi w środowisku naturalnym. Z jednej strony wiemy, że poza laboratorium dany rodzaj organizmu (na przykład E. coli) wykazuje niewiarygodny stopień różnorodności. Z drugiej strony, kiedy sekwencjonujemy genom szczepu hodowanego przez jakiś czas w laboratorium, często obserwujemy tylko niewielką liczbę (kilkaset) zmian związanych z pewną cechą. W przyrodzie sytuacja wygląda zupełnie inaczej. Kilka tygodni temu [z perspektywy 2010 roku] ukazał się mój artykuł, w którym razem ze współautorkami przedstawiliśmy wyniki porównania 61 genomów występujących w środowisku naturalnym, odizolowanych szczepów E. coli. Chociaż niektóre genomy E. coli są dość podobne do siebie nawzajem, to inne są bardzo ODMIENNE – w jednym genomie jest ponad MILION „dodatkowych” zasad (liter DNA), których nie ma w innych genomach15. Ułamek wspólnych białek między dwoma szczepami wynosi od niemal wszystkich (99,7%) do mniej niż połowy (48%). Większość genomów E. coli zawiera około 5000 genów, jeśli jednak przyjrzymy się wszystkim przeanalizowanym dotąd różnym genom, to okazuje się, że istnieje ponad 15 tysięcy różnych rodzin genów (czyli ponad trzy razy więcej niż zawiera dowolny genom E. coli!). Mniej niż tysiąc genów jest utrwalonych we wszystkich zsekwencjonowanych do tej pory genomach E. coli. Co to oznacza? Wybierzmy jakiś genom E. coli i go zsekwencjonujmy. Spośród 5000 genów mniej niż 20% występuje w niemal wszystkich innych genomach E. coli, a dla każdego genu w tym genomie istnieje zapewne dziewięć lub dziesięć genów E. coli występujących w innych genomach E. coli, ale nieobecnych w tym konkretnym genomie E. coli. Szacujemy, że w dodatku do 15 tysięcy odkrytych do tej pory rodzin genów w przewodzie pokarmowym jednej osoby występuje prawdopodobnie około 30 tysięcy więcej rodzin genów E. coli16. Jest to olbrzymia różnorodność informacji genetycznej. Ponieważ te liczne szczepy E. coli mogą łatwo wymieniać między sobą geny i części genów, więc mamy do czynienia z absolutnie ogromnym potencjałem do tworzenia nowych rodzajów białek. Naiwne (szczerze mówiąc) obliczenia Behego nijak mają się do tego potencjału do tworzenia nowych białek i nowych interakcji między białkami. Behe nic o tym potencjale nie wiedział.

Przyjrzyjmy się powyższej ilustracji. Zauważmy, że pospolity laboratoryjny szczep E. coli ma 960 rodzin genów, na podstawie których może utworzyć 4144 rodzaje białek. Są jednak również inne geny, które występują w niektórych genomach E. coli, ale nie ma ich w innych. Jeśli odniesiemy się do przyrody, to można zapytać, ile występuje w niej innych rodzin genów, które zwiększają zasób rodzajów białek E. coli? Szacujemy, że w dodatku do 960 rodzin genów we wspomnianym szczepie laboratoryjnym istnieje około 44 tysięcy rodzin genów, które występują w niektórych genomach E. coli, ale nie w innych.
Chodzi mi więc tutaj o to, że gdy Behe twierdzi, iż w ramach eksperymentów ewolucyjnych z E. coli „zasadniczo nie powstało nic nowego”17, ignoruje on niezmiernie ważne fakty. Ponieważ większość ludzi nie zna odpowiedniej literatury, więc należy uznać, że twierdzenie Behego jest zwodnicze. Istnieje ogromna liczba publikacji, w których pokazuje się, czego może dokonać ewolucja! To oczywiste, że E. coli może ewoluować – i to w dużych skalach – i we właściwych warunkach laboratoryjnych jesteśmy w stanie to zaobserwować na własne oczy. Według mnie nie jest uczciwe branie pod uwagę wyłącznie pierwszej połowy eksperymentów Lenskiego, w których zmian było niewiele, i wyciąganie na tej podstawie wniosku, że E. coli nie ewoluuje. Szczep mutatora (który powstał na półmetku eksperymentów Lenskiego) diametralnie zmienił postać rzeczy.
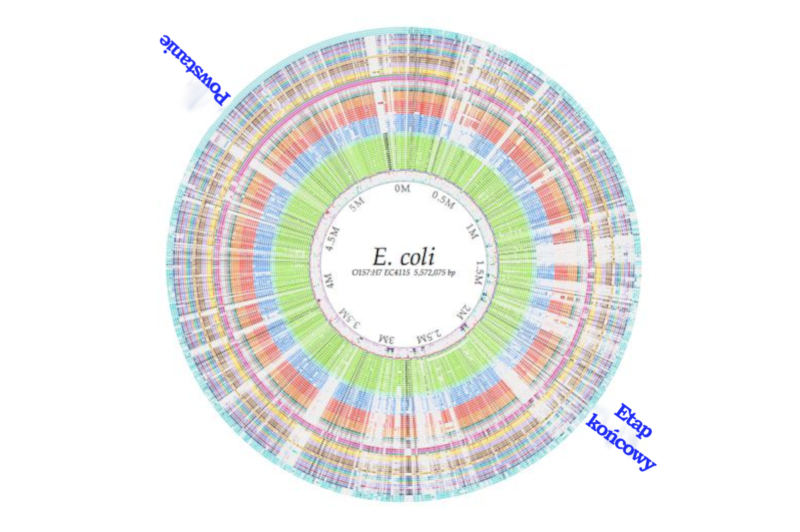 Powyższa ilustracja przedstawia wyniki porównania 61 genomów E. coli (każde koncentryczne koło odpowiada jednemu szczepowi E. coli) ukazujące utrwalenie genów18. Chciałbym zwrócić uwagę na to, że istnieje wiele dużych luk – jaśniej pokolorowane regiony genów, których nie ma w wielu genomach, ale są obecne w innych. Niektóre z tych regionów kodują nowe „maszyny molekularne” – w odróżnieniu od Behego, sądzę, że wiele z nich można nazwać „fundamentalnie nowymi” kompleksami.
Powyższa ilustracja przedstawia wyniki porównania 61 genomów E. coli (każde koncentryczne koło odpowiada jednemu szczepowi E. coli) ukazujące utrwalenie genów18. Chciałbym zwrócić uwagę na to, że istnieje wiele dużych luk – jaśniej pokolorowane regiony genów, których nie ma w wielu genomach, ale są obecne w innych. Niektóre z tych regionów kodują nowe „maszyny molekularne” – w odróżnieniu od Behego, sądzę, że wiele z nich można nazwać „fundamentalnie nowymi” kompleksami.
Oryginał: Is There an Edge to Evolution?, „BioLogos” 2010, December 4 [dostęp: 1 IV 2025].
Przekład z języka angielskiego: Dariusz Sagan
Przypisy
- Por. też części 1–3: D. Ussery, Czy istnieje granica ewolucji? Analiza książki Michaela J. Behego „Granica ewolucji” – rozdział 1, tłum. D. Sagan, „W Poszukiwaniu Projektu” 2025, 26 marca [dostęp: 3 IV 2025]; tenże, Czy istnieje granica ewolucji? Rozdziały 2 i 3 (analiza), tłum. D. Sagan, „W Poszukiwaniu Projektu” 2025, 28 marca [dostęp: 3 IV 2025]; tenże, Czy istnieje granica ewolucji? Rozdziały 4 i 5 (analiza), tłum. D. Sagan, „W Poszukiwaniu Projektu” 2025, 2 kwietnia [dostęp: 3 IV 2025] (przyp. tłum.).
- Por. M.J. Behe, Granica ewolucji. W poszukiwaniu ograniczeń darwinizmu, tłum. Z. Kościuk, „Seria Inteligentny Projekt”, Fundacja En Arche, Warszawa 2020 (przyp. tłum.).
- Tamże, s. 120 (przyp. tłum.).
- Por. R. Dawkins, Wspinaczka na szczyt nieprawdopodobieństwa, tłum. M. Yamazaki, „Na Ścieżkach Nauki”, Prószyński i S-ka, Warszawa 1998 (przyp. tłum.).
- Por. M.J. Behe, Czarna skrzynka Darwina. Biochemiczne wyzwanie dla ewolucjonizmu, tłum. D. Sagan, „Seria Inteligentny Projekt”, Fundacja En Arche, Warszawa 2020 (przyp. tłum.).
- R. Dawkins, Inferior Design, „New York Times” 2007, July 1, https://www.nytimes.com/2007/07/01/books/review/Dawkins-t.html [dostęp: 3 IV 2025].
- M.J. Behe, Granica ewolucji, s. 136–137.
- Por. T.R. Gregory, All About Eye Evolution, „Science 2.0” 2008, December 6 [dostęp: 3 IV 2025].
- Por. np. M.F. Land, D.-E. Nilsson, Animal Eyes, „Oxford Animal Biology Series”, Oxford University Press, Oxford 2002.
- Por. C. Heinis, K. Johnsson, Using Peptide Loop Insertion Mutagenesis for the Evolution of Proteins, „Methods in Molecular Biology” 2010, Vol. 634, s. 217–232, https://doi.org/10.1007/978-1-60761-652-8_16 (przyp. tłum.).
- Por. J. Martin, Beauty Is in the Eye of the Beholder: Proteins Can Recognize Binding Sites of Homologous Proteins in More Than One Way, „PLoS Computational Biology” 2010, Vol. 6, No. 6, numer artykułu: e1000821, https://doi.org/10.1371/journal.pcbi.1000821 (przyp. tłum.).
- Por. też T.R. Gregory, All About Eye Evolution.
- M.J. Behe, Granica ewolucji, s. 163 (przyp. tłum.).
- Por. J.E. Barrick, R.E. Lenski, Genome-Wide Mutational Diversity in an Evolving Population of Escherichia coli, „Cold Spring Harbor Symposia on Quantitative Biology” 2009, Vol. 74, s. 119–129, https://doi.org/10.1101/sqb.2009.74.018.
- Por. O. Lukjancenko, T.M. Wassenaar, D.W. Ussery, Comparison of 61 Sequenced Escherichia coli Genomes, „Microbial Ecology” 2010, Vol. 60, No. 4, s. 708–720, https://doi.org/10.1007/s00248-010-9717-3.
- Por. L. Snipen, T. Almøy, D.W. Ussery, Microbial Comparative Pan-Genomics Using Binomial Mixture Models, „BMC Genomics” 2009, Vol. 10, numer artykułu: 385, https://doi.org/10.1186/1471-2164-10-385.
- M.J. Behe, Granica ewolucji, s. 163.
- Więcej szczegółów por. na ilustracji 5 w: O. Lukjancenko, T.M. Wassenaar, D.W. Ussery, Comparison of 61 Sequenced Escherichia coli Genomes.
Literatura:
1. Barrick J.E., Lenski R.E., Genome-Wide Mutational Diversity in an Evolving Population of Escherichia coli, „Cold Spring Harbor Symposia on Quantitative Biology” 2009, Vol. 74, s. 119–129, https://doi.org/10.1101/sqb.2009.74.018.
2. Behe M.J., Czarna skrzynka Darwina. Biochemiczne wyzwanie dla ewolucjonizmu, tłum. D. Sagan, „Seria Inteligentny Projekt”, Fundacja En Arche, Warszawa 2020.
3. Behe M.J., Granica ewolucji. W poszukiwaniu ograniczeń darwinizmu, tłum. Z. Kościuk, „Seria Inteligentny Projekt”, Fundacja En Arche, Warszawa 2020.
4. Dawkins R., Inferior Design, „New York Times” 2007, July 1 [dostęp: 3 IV 2025].
5. Dawkins R., Wspinaczka na szczyt nieprawdopodobieństwa, tłum. M. Yamazaki, „Na Ścieżkach Nauki”, Prószyński i S-ka, Warszawa 1998.
6. Gregory T.R., All About Eye Evolution, „Science 2.0” 2008, December 6 [dostęp: 3 IV 2025].
7. Heinis C., Johnsson K., Using Peptide Loop Insertion Mutagenesis for the Evolution of Proteins, „Methods in Molecular Biology” 2010, Vol. 634, s. 217–232, https://doi.org/10.1007/978-1-60761-652-8_16.
8. Land M.F., Nilsson D.-E., Animal Eyes, „Oxford Animal Biology Series”, Oxford University Press, Oxford 2002.
9. Lukjancenko O., Wassenaar T.M., Ussery D.W., Comparison of 61 Sequenced Escherichia coli Genomes, „Microbial Ecology” 2010, Vol. 60, No. 4, s. 708–720, https://doi.org/10.1007/s00248-010-9717-3.
10. Martin J., Beauty Is in the Eye of the Beholder: Proteins Can Recognize Binding Sites of Homologous Proteins in More Than One Way, „PLoS Computational Biology” 2010, Vol. 6, No. 6, numer artykułu: e1000821, https://doi.org/10.1371/journal.pcbi.1000821.
11. Snipen L., Almøy T., Ussery D.W., Microbial Comparative Pan-Genomics Using Binomial Mixture Models, „BMC Genomics” 2009, Vol. 10, numer artykułu: 385, https://doi.org/10.1186/1471-2164-10-385.
12. Ussery D., Czy istnieje granica ewolucji? Analiza książki Michaela J. Behego „Granica ewolucji” – rozdział 1, tłum. D. Sagan, „W Poszukiwaniu Projektu” 2025, 26 marca [dostęp: 3 IV 2025].
13. Ussery D., Czy istnieje granica ewolucji? Rozdziały 2 i 3 (analiza), tłum. D. Sagan, „W Poszukiwaniu Projektu” 2025, 28 marca [dostęp: 3 IV 2025].
14. Ussery D., Czy istnieje granica ewolucji? Rozdziały 4 i 5 (analiza), tłum. D. Sagan, „W Poszukiwaniu Projektu” 2025, 2 kwietnia [dostęp: 3 IV 2025].

