W przełomowej publikacji w „Nature” donoszono o „kompletnym zsekwencjonowaniu genomów małp człekokształtnych”, obejmującym genomy szympansów zwyczajnych, szympansów karłowatych, goryli nizinnych, orangutanów borneańskich, orangutanów sumatrzańskich oraz siamangów wielkich1. Zwróciłem na to uwagę we wcześniejszym artykule, informując, że jedna z ikon ewolucji – słynna „jednoprocentowa różnica” między genomem człowieka a genomem szympansa, głoszona w licznych publikacjach naukowych oraz popularnonaukowych, a także w dydaktyce – legła w gruzach2. Z jakichś powodów (trudno powiedzieć z jakich, bo nie jestem jasnowidzem) badacze zdecydowali się ukryć to przełomowe odkrycie w gąszczu technicznego żargonu w sekcji Dane uzupełniające. Przejdźmy zatem do szczegółów naukowych.
Być może myślicie sobie: „zaraz, zaraz, czy te genomy nie zostały zsekwencjonowane już dawno temu?”. Odpowiedź brzmi: i tak, i nie. Owszem, już wcześniej podejmowaliśmy próby zsekwencjonowania genomów tych gatunków, ale jak wyjaśniają autorzy artykułu: „ze względu na powtarzalny charakter genomów małp człekokształtnych nie udało się uzyskać kompletnych zestawień genomowych. Dotychczasowe sekwencje referencyjne nie mają wystarczającej rozdzielczości dla niektórych z najbardziej dynamicznych regionów genomu, w tym w obszarach odpowiadających rodzinom genów specyficznych dla danej linii ewolucyjnej”3.
Innymi słowy, jak ujmuje to towarzyszący publikacji artykuł wyjaśniający:
W przeszłości naukowcy potrafili rozszyfrować fragmenty genomów małp człekokształtnych, ale uczonym nigdy nie udało się złożyć kompletnej sekwencji dla żadnego gatunku. W obecnym badaniu jednak [Kateryna] Makova i jej współpracownicy zastosowali zaawansowane techniki sekwencjonowania oraz algorytmy, które umożliwiły odczyt długich odcinków DNA i złożenie ich w sekwencję rozciągającą się od jednego końca chromosomu do drugiego, bez żadnych luk. „Ni-gdy wcześniej tego nie dokonano” – twierdzi Makova .
A zatem kompletne genomy małp człekokształtnych nigdy wcześniej nie zostały w pełni zsekwencjonowane. Co więcej, używano genomu ludzkiego jako sekwencji referencyjnej, co sprawiało, że genomy małp człekokształtnych wydawały się bardziej podobne do ludzkich, niż były w rzeczywistości.
Nie wierzycie?
Jak dowiadujemy się z artykułu:
Większość wcześniejszych badań porównawczych genomów małp człekokształtnych była ograniczana przez konieczność mapowania zestawień o niższej jakości na wysokiej jakości genom człowieka. Skutkowało to powstawaniem systematycznych błędów związanych z wykorzystaniem ludzkiego genomu referencyjnego4.
Jest to zgodne z tym, co w 2007 roku stwierdziło Narodowe Centrum Informacji Biotechnologicznej (NCBI) na temat wczesnej wersji roboczej genomu szympansa:
Kontigi (ang. contigs) składano, używając genomu ludzkiego jako wzorca, dlatego też w swojej konstrukcji są one „humanizowane”5. Jest to istotna różnica, ponieważ niektóre sekwencje, takie jak insercje, delecje czy duplikacje genów, mogą nie być dokładnie odwzorowane w obecnym złożeniu genomu szympansa6.
A zatem do tej pory większość wersji genomów szympansa i innych małp człekokształtnych była de facto „humanizowana”, ponieważ „składano je, używając genomu ludzkiego jako wzorca”7. To w praktyce sprawia, że wydają się one bardziej podobne do genomu ludzkiego, niż są w rzeczywistości. Czy te nowe wersje robocze genomów małp człekokształtnych mogą pomóc rozwiązać ten problem?
Problem rozwiązany?
Inny artykuł wyjaśniający w „Nature” zdaje się sugerować, że te „kompletne” wersje robocze genomów małp człekokształtnych udowodnią, iż są one mniej podobne do genomu ludzkiego, niż dotychczas twierdzono:
Wkrótce po tym, jak w 2003 roku ukończono prace nad pierwszą sekwencją genomu ludzkiego, opublikowano złożenie genomu szympansa. Następnie pojawiły się informacje dotyczące złożeń genomów innych wielkich małp człekokształtnych, takich jak goryl nizinny, orangutan sumatrzański i szympans karłowaty, a także małych małp człekokształtnych – mniej spokrewnionych z człowiekiem niż wielkie małpy człekokształtne. Genomy te stanowiły cenną okazję do skatalogowania nagromadzonych podczas ewolucji małp człekokształtnych różnic genetycznych, włączając w to zmiany unikatowe dla ludzi. Jednak ze względu na niekompletność pierwszych, roboczych wersji genomu porównania ograniczały się wyłącznie do poprawnie rozszyfrowanych fragmentów genomu. Badania te koncentrowały się zatem wyłącznie na stosunkowo niewielkich różnicach i pomijały wysoce powtarzalne sekwencje oraz wielkoskalowe różnice strukturalne, takie jak inwersje czy duplikacje sekwencji genomowych8.
Ostatnie zdanie wydaje się sugerować, że wcześniejsze porównania genomu człowieka i genomów małp człekokształtnych „koncentrowały się wyłącznie na stosunkowo niewielkich różnicach” i „pomijały” fragmenty, które pociągają za sobą „wielkoskalowe różnice strukturalne”9. Autor zauważa, że w ramach tego badania „w pełni zsekwencjonowano genomy sześciu żyjących współcześnie gatunków małp człekokształtnych, co umożliwiło długo oczekiwane porównania trudnych do złożenia regionów genomowych”10. W związku z tym można oczekiwać, że te nowe, „kompletne” genomy małp człekokształtnych ujawnią znacznie większe różnice w porównaniu z genomem człowieka.
Nowe genomy małp człekokształtnych a genom ludzki
Co zastanawiające, podczas lektury artykułu technicznego trudno znaleźć bezpośrednie porównanie genomów małp człekokształtnych i człowieka. Poniższy fragment jest chyba jedynym, który się do tego zbliża:
Ogólnie rzecz biorąc, porównania sekwencji kompletnych genomów małp człekokształtnych wykazały większe rozbieżności niż dotychczas szacowano (Dane uzupełniające III–IV). W istocie od 12,5 do 27,3% sekwencji genomów małp człekokształtnych nie udało się dopasować lub było niezgodne z prostym dopasowaniem jeden do jednego, co skutkowało powstaniem luk11.
Co to dokładnie oznacza? Po pierwsze, naukowcy sami przyznają, że „porównania sekwencji między kompletnymi genomami małp człekokształtnych wykazały większe rozbieżności niż wcześniej szacowano”12. Jednak w publikacji w „Nature” człowiek jest traktowany jako „małpa człekokształtna”, więc w tym stwierdzeniu ukryte jest założenie, że porównywanie „genomów małp człekokształtnych” obejmuje także zestawienia genomu ludzkiego i genomów małp człekokształtnych (tj. innych niż człowiek hominoidów)13. Dlatego w dalszej części tego artykułu będę nazywał ludzi po prostu „ludźmi”, a „inne niż człowiek hominoidy” – „małpami człekokształtnymi”, tak jak robi to większość ludzi.
Co istotne, w ubiegłym roku na „bioRxiv” ukazały się dwie wersje preprintu tej pracy (wersja 1 i 2), które najprawdopodobniej odpowiadają manuskryptowi złożonemu pierwotnie do „Nature” oraz jego wersji po pierwszych recenzjach i naniesionych poprawkach. W obu wersjach wynik ten poprzedzają dwa następujące zdania14:
Często cytowana statystyka o około 99-procentowej identyczności sekwencji genetycznej szympansa i człowieka jest prawdziwa dla większości genomu, gdy weźmie się pod uwagę warianty pojedynczego nukleotydu (SNV). Jednakże porównania genomów T2T sugerują znacznie bardziej precyzyjną ocenę15.
T2T oznacza badanie „od telomeru do telomeru” – czyli analizę całych chromosomów w obrębie genomu. Zdania te zostały najwyraźniej usunięte podczas poprawek do wersji opublikowanej w „Nature”, co jest interesującą decyzją redakcyjną. Co zatem wcześniejsze wersje artykułu mówią o różnicach między człowiekiem a szympansem?
Jak się zaraz przekonamy, powyższe stwierdzenie – że „porównania sekwencji między kompletnymi genomami małp wykazały większe rozbieżności niż wcześniej szacowano”16 – jest prawdziwe. Ale nie ujawnia ono skali różnic między genomem ludzkim a genomami małp człekokształtnych. Przejdźmy więc do sedna.
Spójrzmy powtórnie na te liczby – od 12,5 do 27,3%. Te same wartości pojawiają się ponownie – głęboko ukryte w sekcji Dane uzupełniające, gdzie porównano genomy małp człekokształtnych z genomem człowieka17. Można je odnaleźć, o ile się wie, gdzie ich szukać. Ale czy powinienem powiedzieć „ukryte” – czy raczej „schowane”? Dane te wskazują, że spośród wszystkich małp człekokształtnych genom szympansa jest najbardziej podobny do genomu ludzkiego, wykazując 12,5% „rozbieżności wynikającej z luk” względem genomu ludzkiego! Co więcej, gdy porównanie przeprowadzono w odwrotnym układzie – traktując genom człowieka jako sekwencję docelową, a szympansa jako zapytanie – różnica wyniosła 13,3%. Pozwólcie, że wyrażę się jasno: zgodnie z tym badaniem, genomy człowieka i szympansa nie są identyczne w 98,8% (ani różne w 1,2%) – jak można przeczytać na stronie internetowej Narodowego Muzeum Historii Naturalnej w Waszyngtonie (więcej na ten temat w moim tekście Visitor’s Guide [Przewodnik dla zwiedzających])18. W rzeczywistości są one podobne najwyżej w 87,5%, co oznacza, że genomy człowieka i szympansa różnią się co najmniej o 12,5%, a być może nawet o 13,3%! Właśnie ta różnica 13,3% jest bardziej istotna, ponieważ odzwierciedla stopień podobieństwa genomu człowieka do genomu szympansa.
Czym dokładnie jest „rozbieżność wynikającą z luk”?
Zanim pójdziemy dalej, chcę wyjaśnić, co dokładnie autorzy rozumieją pod pojęciem „rozbieżność wynikająca z luk” (gap divergence) lub „rozbieżność wynikająca z różnic” (gap difference). Artykuł rozbieżność wynikającą z luk definiuje w następujący sposób:
Rozbieżność wynikającą z luk definiuje się jako odsetek pozycji w haplotypie doce-lowym, które nie zostały dopasowane do drugiego haplotypu. Może to wynikać z pro-cesów biologicznych (np. utrata/powstanie genów oraz insercje/delecje), brakujących danych lub problemów technicznych (np. błędu dopasowania z powodu zmian struk-turalnych (SV), elementów powtarzalnych itp.)19.
Jak zatem określa się „rozbieżność wynikającą z luk”? Z tego, co rozumiem, polega to na podzieleniu genomu docelowego w ramach dopasowania genomów na segmenty o długości 1 miliona par zasad (1 Mbp) i sprawdzeniu, ile zasad w każdym takim segmencie nie ma odpowiadającej zasady w zestawionym z nim genomie zapytania. Jeśli cały segment 1 Mbp nie ma żadnego dopasowania, jego rozbieżność luk wynosi 100%. Jeśli 10 000 par zasad nie ma dopasowania, rozbieżność luk wynosi 1%, jeśli 1000 par zasad, to 0,1% itd. Zgodnie z wynikami badania średnia wartość „rozbieżności wynikającej z luk” w każdym segmencie 1 Mbp dopasowanego do genomu szympansa (jako sekwencji zapytania) genomu człowieka (jako sekwencji docelowej) wynosi 12,5%. Innymi słowy, 12,5% zasad w genomie człowieka nie znajduje odpowiedników w genomie szympansa (porównując całe genomy).
Sporządzona wyłącznie w celach poglądowych poniższa ilustracja (nie została ona zaczerpnięta z tekstu opublikowanego w „Nature”) pomoże zobrazować różnice między „wariantami pojedynczego nukleotydu” (SNV) a „lukami” (gaps) w obu genomach:
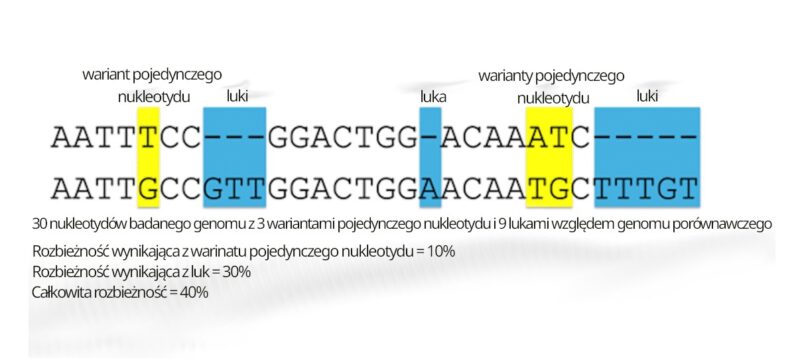
Jak widać, luki reprezentują nukleotydy lub segmenty nukleotydów, które po prostu nie występują w jednym bądź drugim genomie, podczas gdy „warianty pojedynczego nukleotydu” (SNV) występują w różnych formach. Oba rodzaje różnic można zsumować, aby obliczyć całkowitą różnicę między genomami.
Górna granica oszacowania
Dlaczego więc w głównym tekście pojawia się zakres od 12,5 do 27,3%? Dlatego że górna wartość niezgodności między genomem goryla a genomem ludzkim to zdumiewające 27,3%. Jeśli spojrzymy na ilustrację III.12 w sekcji Danych uzupełniających, znajdziemy następujące wartości procentowe „rozbieżności wynikającej z luk” między genomami różnych małp człekokształtnych w porównaniu z genomem ludzkim (porównując tylko chromosomy autosomalne):
- orangutan sumatrzański (Pongo abelii) vs człowiek: 15,4% i 16,5% „rozbieżności wynikającej z luk” (tj. minimalna różnica),
- goryl nizinny (Gorilla gorilla) vs człowiek: 17,9% i 27,3% „rozbieżności wynikającej z luk” (tj. minimalna różnica),
- szympans karłowaty (Pan paniscus) vs człowiek: 12,5% i 14,4% „rozbieżności wynikającej z luk” (tj. minimalna różnica),
- szympans zwyczajny (Pan troglodytes) vs człowiek: 12,5% i 13,3% „rozbieżności wynikającej z luk” (tj. minimalna różnica)20.
Widzicie, jak łatwo jest podsumować te dane? To ogromne odkrycia zarówno dla nauki, jak i kultury popularnej, a jednak artykuł w „Nature” oraz dwa teksty wyjaśniające w tym samym czasopiśmie nie potrafiły jasno ich wyeksponować. Dane te zniknęły w gąszczu technicznego żargonu i mało przejrzystych opisów w sekcji Dane uzupełniające, a zdanie o „często cytowanej statystyce około 99% identyczności sekwencji” zostało usunięte w trakcie poprawek artykułu. „Nature” – o czym mogę z całą pewnością zapewnić – nie jest czasopismem redagowanym na oślep. Były to świadome decyzje podjęte przez kogoś podczas procesu redakcyjnego. Ten brak klarowności jest po prostu niewiarygodny.
Szczegóły techniczne
Głęboko w sekcji Dane uzupełniające znaleźć możemy ilustrację III.12, która wyjaśnia „rozbieżność wynikającą z luk” między różnymi gatunkami:
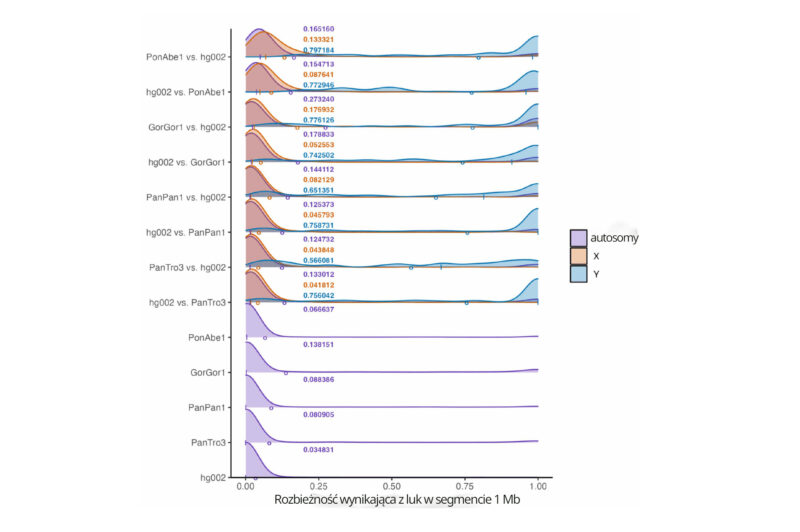
Uwaga autora [C. Luskina] z 8 lipa 2025 roku: Zamieszczam tutaj pełny wykres z ilustracji III.12 z sekcji Dane uzupełniające do pracy Yoo i współpracowników, ponieważ niektórzy twierdzą, że pierwotnie pominąłem dane z wykresu, gdyż w jakiś sposób obalały one moją argumentację. Nie, to nieprawda. Jedyny powód, dla którego zamieściłem tylko porównania genomów ludzkich i małp człekokształtnych (górne osiem krzywych), stanowiła chęć uproszczenia, ponieważ tylko ta część wykresu była istotna dla mojej argumentacji (wewnątrzgatunkowe różnice genetyczne – dolne pięć krzywych – są nieistotne). Moja argumentacja nie polega na tym, że różnice genetyczne między człowiekiem a małpami człekokształtnymi całkowicie obalają teorię ewolucji. W mojej argumentacji chodzi o to, że ewolucjoniści, którzy przez dekady twierdzili, iż ludzie różnią się genetycznie od małp tylko o 1%, mylili się. Górne osiem krzywych bardzo wyraźnie to pokazuje. Na wykresie nie było ani nie ma żadnych danych, które podważałyby mój argument, a mimo to zawsze zamieszczałem odnośnik do sekcji Danych uzupełniających, gdzie każdy mógł mieć dostęp do pełnego zestawu danych21. Nie ma tu nic do ukrycia. Co więcej, już wcześniej – zanim pojawiły się głosy krytyczne – szczegółowo odniosłem się do zastrzeżeń dotyczących wewnątrzgatunkowych różnic genetycznych22.
Podpis pod ilustracją w sekcji Danych uzupełniających brzmi: „Wykresy przedstawiają segmenty o długości 1 Mbp pogrupowane według rozbieżności wynikającej z luk dla każdego dopasowania par sekwencji”, gdzie dopasowanie par sekwencji to próba zestawienia dwóch sekwencji w celu określenia stopnia ich podobieństwa bądź różnicy23. Otrzymujemy zatem bezpośredni pomiar minimalnego stopnia różnicy między dwoma genomami.
Dodanie wariantów pojedynczego nukleotydu (SNV)
Istnieje jednak jeszcze inny rodzaj zmienności między genomami, również zidentyfikowany w tym artykule – pojedyncze zmiany nukleotydowe (nazywane „wariantami pojedynczych nukleotydów” lub czasem „krótkimi wariantami nukleotydowymi”). W sekcji Dane uzupełniające ukryta jest także ilustracja III.11, która przedstawia odnotowany w tym badaniu procentowy udział SNV między genomem człowieka a genomami różnych małp człekokształtnych. Oto, co ustalono:
- orangutan sumatrzański (Pongo abelii) vs człowiek: różnica ok. 3,6%,
- goryl nizinny (Gorilla gorilla) vs człowiek: różnica 1,9–2,0%,
- szympans karłowaty (Pan paniscus) vs człowiek: różnica 1,5–1,6%,
- szympans zwyczajny (Pan troglodytes) vs człowiek: 1,5–1,6% różnicy24.
Jeśli dodamy „rozbieżność wynikającą z luk” do różnic wynikających z SNV, otrzymamy następujący całkowity stopień zróżnicowania między genomem ludzkimi a genomami małp człekokształtnych:
- orangutan sumatrzański (Pongo abelii) vs człowiek: różnica ok. 19,0–20,1%,
- goryl nizinny (Gorilla gorilla) vs człowiek: różnica ok. 19,8–29,3%,
- szympans karłowaty (Pan paniscus) vs człowiek: różnica ok. 14,0–16,0%,
- szympans zwyczajny (Pan troglodytes) (genom docelowy) vs człowiek: różnica ok. 14,0%,
- człowiek (genom docelowy) vs szympans zwyczajny (Pan troglodytes): różnica ok. 14,9%25.
Widzimy zatem, że całkowita różnica między genomem człowieka a genomem szympansa wynosi około 14,9%. Stanowi to znacznie większy stopień zróżnicowania niż często powtarzana statystyka, według której różnimy się genetycznie od szympansów zaledwie o 1%!
Czy to już wszystko?
Niewątpliwie potrzebne są dalsze analizy, aby określić, w jakim stopniu nukleotydy wykazują „dokładne dopasowania jeden do jednego” między genomem człowieka a genomem szympansa, nawet w regionach, które można było łatwiej dopasować. Podejrzewam więc, że stopień różnic między genomami człowieka i szympansa może w przyszłości jeszcze wzrosnąć.
Na razie możemy bezpiecznie stwierdzić, że najnowsze badania wskazują na co najmniej 14,9-procentową różnicę między genomem człowieka a genomem szympansa. Oznacza to, że oba genomy są co najmniej dziesięciokrotnie mniej podobne, niż zwykle się to podaje opinii publicznej.
Oczywiście mówimy tu o 44 chromosomach autosomalnych w genomie ludzkim. Warto również zauważyć, że w porównaniu z szympansami ludzki chromosom Y ma zdumiewającą 56,6-procentową „rozbieżność wynikającą z luk” (oraz 3,9-procentową dywergencję SNV), a ludzki chromosom X ma 4,4-procentową „rozbieżność wynikającą z luk” (oraz 1,1-procentową dywergencję SNV). I to wszystko również ukryto w sekcji Dane uzupełniające.
To odkrycia o przełomowym znaczeniu – tym bardziej godne ubolewania jest to, że redakcja „Nature” nie zaprezentowała tych danych wprost, a ich odnalezienie utrudniono, ukrywając je w technicznym żargonie niezrozumiałym dla większości odbiorców spoza środowiska naukowego. Dlaczego tak się stało? Ważne jest, aby zdać sobie sprawę, że publikowanie artykułów naukowych może przypominać produkcję kiełbasy: jest to często nieapetyczny proces, a ostateczna wersja, którą czytamy, zwykle reprezentuje kompromisowe sformułowania, na których wydrukowanie zgodzili się wszyscy autorzy, recenzenci i redaktorzy – i może nie odzwierciedlać w pełni poglądów każdego autora. Być może niektórzy badacze woleliby przedstawić wnioski bardziej dosadnie. Pozostaje jednak pytanie: dlaczego redakcja „Nature” nie przedstawiła tych wyników w sposób klarowny, pozwalając, by swobodnie dotarły do wszystkich zainteresowanych?
Podejrzewam, że to przełomowe odkrycie będzie miało konsekwencje – nie tylko dla nauki, ale także dla idei wyjątkowości człowieka, wiarygodności szeroko promowanych haseł i wielu innych spraw, o których ludzie będą dyskutować jeszcze przez długi czas. Dla części środowiska naukowego i dziennikarzy naukowych takie rozmowy mogą być niewygodne – szczególnie dla tych, którzy niegdyś głosili dziś już zdyskredytowaną tezę o zaledwie jednoprocentowej różnicy między człowiekiem a szympansem.
Casey Luskin
Oryginał: Fact Check: New “Complete” Chimp Genome Shows 14.9 Percent Difference from Human Genome, „Science & Culture Today” 2025, May 21 [dostęp: 14 I 2026].
Przekład z języka angielskiego: Adam Bechyne
Źródło zdjęcia: Pixabay
Ostatnia aktualizacja strony: 14.1.2026
Przypisy
- Por. D. Yoo et al., Complete Sequencing of Ape Genomes, „Nature” 2025, Vol. 641, No. 8062, s. 401–418, https://doi.org/10.1038/s41586-025-08816-3.
- Por. C. Luskin, Szokujące odkrycie: nowe badania podważają twierdzenie, że różnica między DNA człowieka a DNA szympansa wynosi tylko 1%, tłum. A. Bechyne, „W Poszukiwaniu Projektu” 2026, 9 stycznia [dostęp: 12 I 2026].
- D. Yoo et al., Complete Sequencing of Ape Genomes, s. 401 (przyp. tłum.).
- D, Yoo et al., Complete Sequencing of Ape Genomes, s. 403.
- W genomice i bioinformatyce terminem tym określa się ciągłą sekwencję DNA, zrekonstruowaną na podstawie wielu nakładających się na siebie, krótszych fragmentów (tzw. odczytów) sekwencji DNA, uzyskanych w procesie sekwencjonowania. Kontig reprezentuje spójny, pozbawiony przerw odcinek oryginalnej sekwencji genomowej (przyp. tłum.).
- Chimp Genome Assembly and Annotation Release Notes, „National Center for Biotechnology Information” [dostęp: 26 VIII 2025].
- Tamże (przyp. tłum.).
- L. Kuderna, Complete Ape Genomes Offer a Close-up View of Human Evolution, „Nature” 2025, Vol. 641, No. 8062, s. 313–314, https://doi.org/10.1038/d41586-025-00912-8 [wyróżnienia dodano].
- Tamże (przyp. tłum.).
- Tamże (przyp. tłum.).
- D. Yoo et al., Complete Sequencing of Ape Genomes, s. 403.
- Tamże (przyp. tłum.).
- Por. Człekokształtne, „Wikipedia” [dostęp: 25 VIII 2025].
- Por. D. Yoo et al., Complete Sequencing of Ape Genomes, „bioRxiv” 2024, July 31, https://doi.org/10.1101/2024.07.31.605654; tenże, Complete Sequencing of Ape Genomes, „bioRxiv” 2024, October 5, https://doi.org/10.1101/2024.07.31.605654.
- Tamże, s. 9.
- Tamże (przyp. tłum.).
- Por. D. Yoo et al., Complete Sequencing of Ape Genomes [Supplementary Information], s. 25.
- Por. C. Luskin, Visitor’s Guide: At Nation’s Natural History Museum, Misinformation on Human Origins, and More, „Science & Culture Today” 2023, October 5 [dostęp: 25 VIII 2025].
- D. Yoo et al., Complete Sequencing of Ape Genomes [Supplementary Information], s. 25.
- D. Yoo et al., Complete Sequencing of Ape Genomes [Supplementary Information] (przyp. tłum.).
- Por. tamże.
- Por. C. Luskin, Critics Change the Topic: Do Human-Human Genetic Differences Matter?, „Science & Culture Today” 2025, June 26 [dostęp: 25 VIII 2025].
- D. Yoo et al., Complete Sequencing of Ape Genomes [Supplementary Information], s. 26.
- Tamże (przyp. tłum.).
- Tamże (przyp. tłum.).
Literatura:
1. Basilio H., What Makes Us Human? Milestone Ape Genomes Promise Clues, „Nature” 2025, Vol. 640, 8059, s. 582, https://doi.org/10.1038/d41586-025-01079-y.
2. Chimp Genome Assembly and Annotation Release Notes, „National Center for Biotechnology Information” [dostęp: 26 VIII 2025].
3. Człekokształtne, „Wikipedia” [dostęp: 25 VIII 2025].
4. Kuderna L., Complete Ape Genomes Offer a Close-up View of Human Evolution, „Nature” 2025, Vol. 641, No. 8062, s. 313–314, https://doi.org/10.1038/d41586-025-00912-8.
5. Luskin C., Critics Change the Topic: Do Human-Human Genetic Differences Matter?, „Science & Culture Today” 2025, June 26 [dostęp: 25 VIII 2025].
6. Luskin C., Fact Check: New “Complete” Chimp Genome Shows 14.9 Percent Difference from Human Genome, „Science & Culture Today” 2025, May 21 [dostęp: 25 VIII 2025].
7. Luskin C., Szokujące odkrycie: nowe badania podważają twierdzenie, że różnica między DNA człowieka a DNA szympansa wynosi tylko 1%, tłum. A. Bechyne, „W Poszukiwaniu Projektu” 2026, 9 stycznia [dostęp: 12 I 2026].
8. Luskin C., Visitor’s Guide: At Nation’s Natural History Museum, Misinformation on Human Origins, and More, „Science & Culture Today” 2023, October 5 [dostęp: 25 VIII 2025].
9. Yoo D. et al., Complete Sequencing of Ape Genomes [Supplementary Information] [dostęp: 25 VIII 2025].
10. Yoo D. et al., Complete Sequencing of Ape Genomes, „bioRxiv” 2024, July 31, https://doi.org/10.1101/2024.07.31.605654.
11. Yoo D. et al., Complete Sequencing of Ape Genomes, „bioRxiv” 2024, October 5, https://doi.org/10.1101/2024.07.31.605654.
12. Yoo D. et al., Complete Sequencing of Ape Genomes, „Nature” 2025, Vol. 641, No. 8062, s. 401–418, https://doi.org/10.1038/s41586-025-08816-3.

