We wcześniejszym artykule omówiłem kwestię nieredukowalnej złożoności maszynerii podziału komórkowego u eukariontów1. Tym, co czyni kwestię powstania cyklu komórkowego u eukariontów problemem szczególnie trudnym dla wyjaśnień ewolucjonistycznych, jest to, że między mechanizmem podziału komórkowego u eukariontów a mechanizmem wykorzystywanym przez komórki prokariotyczne istnieje ogromna przepaść – zarówno pod względem zaangażowanych elementów białkowych, jak i pod względem logiki tych mechanizmów. Te dwa układy zasadniczo nie mają nic ze sobą wspólnego. Jak zauważyłem w artykule opublikowanym na łamach czasopisma „BIO-Complexity:
W procesie wklęśnięcia wewnętrznej błony komórki bakteryjnej pośredniczy FtsZ i inne białka składające się na diwisom (divisome). Natomiast w komórkach eukariotycznych z filamentów aktyny i białek motorycznych nazywanych miozynami tworzy się pierścień kurczliwy, który ściska błonę komórkową, tworząc w ten sposób dwie komórki potomne. Mechanizmy segregacji DNA u prokariontów również znacząco różnią się od sposobu segregacji materiału genetycznego u eukariontów. W trakcie mitozy u eukariontów […] zreplikowany DNA komórkowy ulega kondensacji w odrębne chromosomy. Chromosomy ulegają następnie równemu podziałowi i rozdzieleniu do dwóch komórek potomnych za sprawą procesu kierowanego przez wrzeciono podziałowe, co gwarantuje, że każda komórka otrzyma kompletny i identyczny zestaw informacji genetycznej. Mechanizmy odpowiadające za te procesy są więc zupełnie odmienne u prokariontów i eukariontów2.
Zawarta w tym artykule tabela 1 wskazuje znaczące różnice między tymi dwoma mechanizmami podziału komórkowego3.
Bakteryjny proces podziału komórkowego jest nieredukowalnie złożony
Z przeglądem mechanizmów biorących udział w bakteryjnym procesie podziału komórkowego można zapoznać się w dwóch artykułach, które opublikowałem wcześniej na blogu „Evolution News & Science Today”4. Różne cechy maszynerii podziału komórkowego u prokariontów – podobnie do podziału komórkowego u eukariontów – są nieredukowalnie złożone. Na przykład u bakterii Gram-ujemnych do prawidłowego zajścia podziału komórkowego niezbędnych jest minimalnie dziesięć rodzajów białek (FtsA, B, I, K, L, N, Q, W, Z oraz ZipA) i dlatego wskazano je jako potencjalne cele dla antybiotyków5. W celu zaoszczędzenia miejsca zainteresowanych czytelników odsyłam do moich artykułów, w których szczegółowo omówiłem kwestię nieredukowalnej złożoności maszynerii podziału komórkowego u prokariontów.
Złożoność cyklu komórkowego u ostatniego eukariotycznego wspólnego przodka była podobna do współczesnej
Wyniki analizy filostratygraficznej wskazują, że większość elementów występujących w procesie podziału komórkowego u współczesnych eukariontów była już obecna u ostatniego eukariotycznego wspólnego przodka (LECA – last eukaryotic common ancestor). Na przykład w ramach jednego badania wykazano, że minimalnie 24 z 37 znanych podjednostek, koaktywatorów i bezpośrednich lub pośrednich substratów APC/C (anaphase promoting complex/cyclosome) występowała już u LECA6. Podobną analizę przeprowadzono w odniesieniu do elementów punktu kontrolnego związanego z mitozą oraz powiązanych z nimi funkcjonalnych domen i motywów. Badacze doszli do następującego wniosku: „większość elementów punktu kontrolnego jest pradawna i prawdopodobnie była już obecna u ostatniego eukariotycznego wspólnego przodka”7. W innym badaniu potwierdzono ponadto, że kompleks dynaktyny (aktywator cytoplazmatycznej dyneiny, który odgrywa kluczową rolę w procesie mitozy) także jest pradawny i prawdopodobnie wszystkie jego podjednostki występowały już u LECA8. W ramach jeszcze innego badania, w którym przeanalizowano dziewięćdziesiąt różnych eukariotycznych linii rodowych, z wykorzystaniem metody nazywanej parsymonią Dolla, ustalono historie ewolucyjne białek odgrywających rolę w sieci kinetochorów (w tej metodzie zakłada się powstanie co najwyżej jednego nowego białka i przy założeniu maksymalnej parsymonii wnioskuje się o późniejszych utratach tego białka)9. Badacze odkryli, że 49 z 70 rodzajów białek było już obecnych u LECA.
Zważywszy na to, że LECA najwyraźniej miał już większość elementów cyklu komórkowego, należy zapytać, skąd te elementy się wzięły – zwłaszcza jeśli weźmiemy pod uwagę, że mechanizmy podziału komórkowego u eukariontów i prokariontów tak skrajnie się od siebie różnią. Jak już wspomniałem, praktycznie rzecz biorąc nie mają one ze sobą nic wspólnego – zarówno pod względem elementów białkowych, jak i pod względem logiki tych mechanizmów.
Elementy cyklu komórkowego u eukariontów nie mają prokariotycznych homologów
W artykule, który opublikowałem ostatnio w czasopiśmie „BIO-Complexity”, podjąłem próbę ustalenia (przy wykorzystaniu BLAST i innych technik bioinformatycznych) stopnia, w jakim da się zidentyfikować u prokariontów dalekie homologi elementów cyklu komórkowego u eukariontów10 – zwłaszcza u archeonów Asgard, nadtypu archeonów, które uznaje się za najbliższych żyjących krewniaków eukariontów11. Okazało się, że dla ogromnej większości elementów cyklu komórkowego u eukariontów nie da się zidentyfikować prokariotycznych homologów, wliczając w to archeony Asgard. Zamieszczona niżej ilustracja (jest to ilustracja 2 w moim artykule w „BIO-Complexity”) przedstawia moje ustalenia dotyczące elementów kompleksu promującego anafazę lub cyklosomu (APC/C) oraz jego bezpośrednich lub pośrednich celów, punktu kontrolnego związanego z mitozą oraz sieci kinetochorów (dzięki analizie filostratygraficznej wykazano, że wszystkie te elementy były już obecne u LECA).
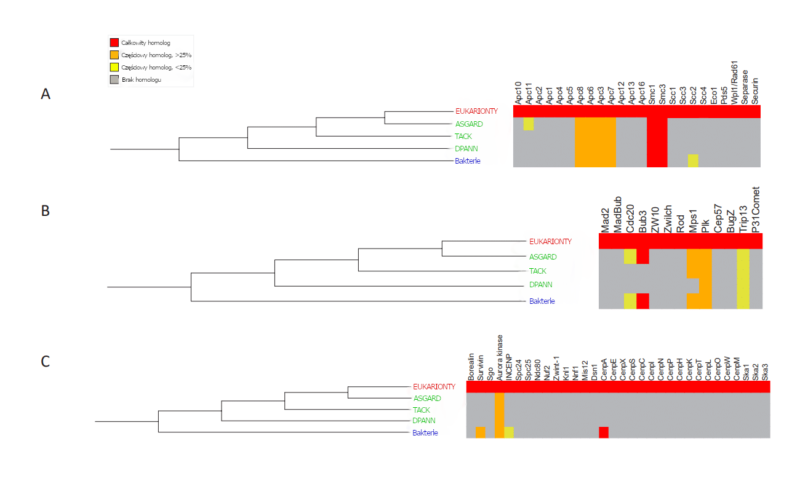
Jak pokazano na ilustracji, ogromna większość białek biorących udział w cyklu komórkowym u eukariontów nie ma żadnych homologów. W przypadkach, w których da się identyfikować homologi, zazwyczaj tylko część białka wykazuje homologiczność (na przykład domena kinazowa kinazy Aurora jest ewidentnie homologiczna do innych kinaz).
Problem dla teorii ewolucji
Jak zauważyłem w artykule opublikowanym w czasopiśmie „BIO-Complexity”:
Te wyniki przemawiają na rzecz hipotezy, zgodnie z którą elementy biorące udział w procesie podziału komórkowego u eukariontów są w znacznej mierze eukariotycznymi innowacjami powstałymi jakiś czas po rozdzieleniu się eukariontów od linii rodowych archeonów. Na pierwszy rzut oka nic nie świadczy o tym, że bardzo odmienne maszynerie replikacji komórek w tych dwóch typach organizmów powstały drogą dziedziczenia z modyfikacjami. To, że na podstawie analizy filostratygraficznej ustalono, iż większość elementów była już obecna u LECA (który zgodnie z szacunkami żył między 1,1 a 2,3 miliarda lat temu), wskazuje, że wszystkie białka eukariontów związane z procesem podziału komórkowego powstały jakiś czas po oddzieleniu się eukariontów od archeonów, ale przed pojawieniem się LECA12.
Co więcej:
W okienku czasowym dostępnym dla powstania systemu kontroli podziału komórkowego u eukariontów wiele białek nie tylko musiało wykształcić swoje specyficzne struktury, tak aby mogły uczestniczyć w cyklu komórkowym, ale musiały też one zastąpić białka biorące udział w bakteryjnym procesie podziału komórkowego oraz utworzyć wysoce skoordynowany układ – i to zachowując przy tym integralność podziału komórkowego oraz procesu segregacji DNA13.
Takie przejście wydaje się szczególnie niewiarygodne, zważywszy na nieredukowalną złożoność zarówno układu prokariotycznego, jak układu eukariotycznego. Każdy element procesu podziału komórkowego u prokariontów nie tylko musiałby zostać zastąpiony, ale ponadto większość białek, które zastępowałyby te elementy, musiałoby powstać de novo. Nawet te kilka białek, które mają homologi u prokariontów, musiałoby pełnić inne funkcje, ponieważ wykonują zupełnie inne zadania w obu układach. Na przykład u prokariontów FtsZ (homolog tubuliny) gromadzi się, by utworzyć pierścień kurczliwy ułatwiający rozdzielenie komórki rodzicielskiej na dwie komórki potomne, a jego eukariotyczny homolog tubulina (podjednostka mikrotubul) odgrywa ważną rolę w segregacji chromosomów w trakcie mitozy.
Przyczyna obdarzona dalekowzrocznością
Jeśli procesy niekierowane są niezdolne do wytworzenia złożonej maszynerii związanej z mitotycznym podziałem komórkowym, to czy istnieje jakaś inna przyczyna, która to potrafi? Jak wyjaśniam w artykule opublikowanym na łamach czasopisma „BIO-Complexity”:
Przejście od prokariontów do eukariontów jest nierozerwalnie związane ze stworzeniem nowej informacji w formie genów koniecznych do kodowania ekspresji licznych potrzebnych białek (których większość jest nieobecna u prokariontów). Ponadto funkcje tych białek muszą być ściśle regulowane i kontrolowane przez różne mechanizmy punktów kontrolnych. Jeszcze poważniejszym problemem dla procesów standardowo przywoływanych w biologii ewolucyjnej jest to, że interesujące nas tutaj przejście musiałoby nastąpić wieloma małymi, stale przyrastającymi krokami, z których każdy zapewnia jakąś funkcjonalną przewagę, utrzymując jednocześnie integralność procesu podziału komórkowego. Jak jednak się przekonaliśmy, wiele niezbędnych procesów jest nieredukowalnie złożonych, co oznacza, że wymagane byłyby liczne, wzajemnie współzależne zmiany, zanim udałoby się uzyskać wyższy stopień przystosowania.
Wiemy z doświadczenia, że istoty inteligentne są zdolne do szybkiego wprowadzania nowej informacji do układu w celu dokonania radykalnej zmiany jego podstawowych składników i utworzenia nowego zespołu zintegrowanych części, które pełnią jakąś funkcję. W każdym innym obszarze doświadczenia zwykle przypisujemy takie inżynieryjne układy sprawczości inteligentnej – przyczynie obdarzonej dalekowzrocznością i potrafiącej obmyślać plany na przyszłość, wizualizować złożone cele i świadomie łączyć ze sobą wszystko, co jest potrzebne do zrealizowania jakiegoś złożonego celu14.
Skrajna różnica między maszynerią podziału komórkowego u eukariontów i prokariontów jest niezmiernie zaskakująca w świetle standardowej ewolucjonistycznej wizji stopniowej, przyrostowej ewolucji. Ta różnica jest natomiast znacznie mniej zaskakująca z perspektywy hipotezy projektu. Zgromadzone dane w większym stopniu potwierdzają ujęcie teleologiczne niż ewolucjonistyczne.
Jonathan McLatchie
Oryginał: The Cell Division Challenge to Eukaryogenesis – And to Evolution, „Evolution News & Science Today” 2024, December 24 [dostęp: 20 VIII 2025].
Przekład z języka angielskiego: Dariusz Sagan
Przypisy
- Por. J. McLatchie, Cykl komórkowy u eukariontów jest układem nieredukowalnie złożonym, tłum. D. Sagan, „W Poszukiwaniu Projektu” 2025, 28 maja [dostęp: 12 VIII 2025].
- Por. tenże, Phylogenetic Challenges to the Evolutionary Origin of the Eukaryotic Cell Cycle, „BIO-Complexity” 2024, No. 4, s. 8 [1–19], https://doi.org/10.5048/BIO-C.2024.4.
- Por. tamże, s. 9–10.
- Por. tenże, Irreducibly Complex, Bacterial Cell Wall Manufacture Is an Evolutionary Enigma, „Evolution News & Science Today” 2023, July 25 [dostęp: 15 IV 2025]; tenże, Bacterial Cell Division and Peptidoglycan Synthesis: An Evolutionary Enigma, „Evolution News & Science Today” 2013, April 3 [dostęp: 15 IV 2025].
- Por. T. den Blaauwen, J.M. Andreu, O. Monasterio, Bacterial Cell Division Proteins as Antibiotic Targets, „Bioorganic Chemistry” 2014, Vol. 55, s. 27–38, https://doi.org/10.1016/j.bioorg.2014.03.007; R.L. Lock, E.J. Harry, Cell-Division Inhibitors: New Insights for Future Antibiotics, „Nature Reviews Drug Discovery” 2008, Vol. 7, No. 4, s. 324–338, https://doi.org/10.1038/nrd2510.
- Por. L. Eme et al., The Phylogenomic Analysis of the Anaphase Promoting Complex and Its Targets Points to Complex and Modern-Like Control of the Cell Cycle in the Last Common Ancestor of Eukaryotes, „BMC Evolutionary Biology” 2011, Vol. 11, numer artykułu: 265, https://doi.org/10.1186/1471-2148-11-265.
- M. Vleugel et al., Evolution and Function of the Mitotic Checkpoint, „Developmental Cell” 2012, Vol. 23, No. 2, s. 240 [239–250], https://doi.org/10.1016/j.devcel.2012.06.013.
- Por. B. Hammesfahr, M. Kollmar, Evolution of the Eukaryotic Dynactin Complex, the Activator of Cytoplasmic Dynein, „BMC Evolutionary Biology” 2012, Vol. 12, numer artykułu: 95, https://doi.org/10.1186/1471-2148-12-95.
- Por. J.J.E. van Hoof et al., Evolutionary Dynamics of the Kinetochore Network in Eukaryotes as Revealed by Comparative Genomics, „EMBO Reports” 2017, Vol. 18, No. 9, s. 1559–1571, https://doi.org/10.15252/embr.201744102.
- Por. J. McLatchie, Phylogenetic Challenges to the Evolutionary Origin of the Eukaryotic Cell Cycle. Por. też K. Zaremba-Niedźwiedzka et al., Asgard Archaea Illuminate the Origin of Eukaryotic Cellular Complexity, „Nature” 2017, Vol. 541, No. 7637, s. 353–358, https://doi.org/10.1038/nature21031.
- Por. A. Spang et al., Complex Archaea That Bridge the Gap between Prokaryotes and Eukaryotes, „Nature” 2015, Vol. 521, No. 7551, s. 173–179, https://doi.org/10.1038/nature14447.
- J. McLatchie, Phylogenetic Challenges to the Evolutionary Origin of the Eukaryotic Cell Cycle, s. 12.
- Tamże.
- Tamże, s. 13 (przyp. tłum.).
Literatura:
1. den Blaauwen T., Andreu J.M., Monasterio O., Bacterial Cell Division Proteins as Antibiotic Targets, „Bioorganic Chemistry” 2014, Vol. 55, s. 27–38, https://doi.org/10.1016/j.bioorg.2014.03.007.
2. Eme L. et al., The Phylogenomic Analysis of the Anaphase Promoting Complex and Its Targets Points to Complex and Modern-Like Control of the Cell Cycle in the Last Common Ancestor of Eukaryotes, „BMC Evolutionary Biology” 2011, Vol. 11, numer artykułu: 265, https://doi.org/10.1186/1471-2148-11-265.
3. Hammesfahr B., Kollmar M., Evolution of the Eukaryotic Dynactin Complex, the Activator of Cytoplasmic Dynein, „BMC Evolutionary Biology” 2012, Vol. 12, numer artykułu: 95, https://doi.org/10.1186/1471-2148-12-95.
4. Lock R.L., Harry E.J., Cell-Division Inhibitors: New Insights for Future Antibiotics, „Nature Reviews Drug Discovery” 2008, Vol. 7, No. 4, s. 324–338, https://doi.org/10.1038/nrd2510.
5. McLatchie J., Bacterial Cell Division and Peptidoglycan Synthesis: An Evolutionary Enigma, „Evolution News & Science Today” 2013, April 3 [dostęp: 15 IV 2025].
6. McLatchie J., Cykl komórkowy u eukariontów jest układem nieredukowalnie złożonym, tłum. D. Sagan, „W Poszukiwaniu Projektu” 2025, 28 maja [dostęp: 12 VIII 2025].
7. McLatchie J., Irreducibly Complex, Bacterial Cell Wall Manufacture Is an Evolutionary Enigma, „Evolution News & Science Today” 2023, July 25 [dostęp: 15 IV 2025].
8. McLatchie J., Phylogenetic Challenges to the Evolutionary Origin of the Eukaryotic Cell Cycle, „BIO-Complexity” 2024, No. 4, s. 1–19, https://doi.org/10.5048/BIO-C.2024.4.
9. McLatchie J., Phylogenetic Challenges to the Evolutionary Origin of the Eukaryotic Cell Cycle; K. Zaremba-Niedźwiedzka et al., Asgard Archaea Illuminate the Origin of Eukaryotic Cellular Complexity, „Nature” 2017, Vol. 541, No. 7637, s. 353–358, https://doi.org/10.1038/nature21031.
10. Spang A. et al., Complex Archaea That Bridge the Gap between Prokaryotes and Eukaryotes, „Nature” 2015, Vol. 521, No. 7551, s. 173–179, https://doi.org/10.1038/nature14447.
11. van Hoof J.J.E. et al., Evolutionary Dynamics of the Kinetochore Network in Eukaryotes as Revealed by Comparative Genomics, „EMBO Reports” 2017, Vol. 18, No. 9, s. 1559–1571, https://doi.org/10.15252/embr.201744102.
12. Vleugel M. et al., Evolution and Function of the Mitotic Checkpoint, „Developmental Cell” 2012, Vol. 23, No. 2, s. 239–250, https://doi.org/10.1016/j.devcel.2012.06.013.

